# 安装包
if (!requireNamespace("dplyr", quietly = TRUE)) {
install.packages("dplyr")
}
if (!requireNamespace("ggbiplot", quietly = TRUE)) {
install.packages("ggbiplot")
}
# 加载包
library(ggbiplot)
library(dplyr)载荷图
示例
载荷图(Biplot)是一种可视化工具,用于展示主成分分析(principal component analysis,PCA)的结果。它可以同时显示数据集中的样本和变量在主成分空间中的位置。在二维Biplot图中,样本通常以点的形式表示,而变量则以箭头表示,箭头的方向和长度分别代表了变量对主成分的贡献度和相关性。
环境配置
系统要求: 跨平台(Linux/MacOS/Windows)
编程语言:R
依赖包:
dplyr,ggbiplot
sessioninfo::session_info("attached")─ Session info ───────────────────────────────────────────────────────────────
setting value
version R version 4.5.2 (2025-10-31)
os Ubuntu 24.04.3 LTS
system x86_64, linux-gnu
ui X11
language (EN)
collate C.UTF-8
ctype C.UTF-8
tz UTC
date 2026-02-04
pandoc 3.1.3 @ /usr/bin/ (via rmarkdown)
quarto 1.8.27 @ /usr/local/bin/quarto
─ Packages ───────────────────────────────────────────────────────────────────
package * version date (UTC) lib source
dplyr * 1.1.4 2023-11-17 [1] RSPM
ggbiplot * 0.6.2 2024-01-08 [1] RSPM
ggplot2 * 4.0.1 2025-11-14 [1] RSPM
[1] /home/runner/work/_temp/Library
[2] /opt/R/4.5.2/lib/R/site-library
[3] /opt/R/4.5.2/lib/R/library
* ── Packages attached to the search path.
──────────────────────────────────────────────────────────────────────────────数据准备
数据来源于R自带的iris数据集。
data("iris")
head(iris) Sepal.Length Sepal.Width Petal.Length Petal.Width Species
1 5.1 3.5 1.4 0.2 setosa
2 4.9 3.0 1.4 0.2 setosa
3 4.7 3.2 1.3 0.2 setosa
4 4.6 3.1 1.5 0.2 setosa
5 5.0 3.6 1.4 0.2 setosa
6 5.4 3.9 1.7 0.4 setosa可视化
通过prcomp函数进行主成分分析。
iris.pca <- prcomp (~ Sepal.Length + Sepal.Width + Petal.Length + Petal.Width,
data=iris,
scale. = TRUE)
summary(iris.pca)Importance of components:
PC1 PC2 PC3 PC4
Standard deviation 1.7084 0.9560 0.38309 0.14393
Proportion of Variance 0.7296 0.2285 0.03669 0.00518
Cumulative Proportion 0.7296 0.9581 0.99482 1.000001. 基础绘图
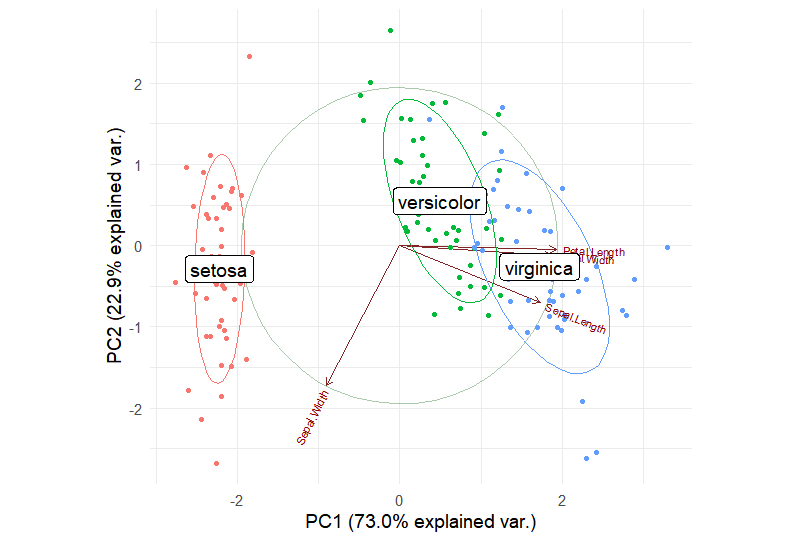
通过ggbipolot绘制PCA结果的载荷图。
iris.gg <-
ggbiplot(iris.pca, obs.scale = 1, var.scale = 1,
groups = iris$Species, point.size=2,
varname.size = 3,
varname.color = "black",
varname.adjust = 1.2,
ellipse = TRUE,
circle = TRUE) +
labs(fill = "Species", color = "Species") +
theme_minimal(base_size = 14) +
theme(legend.direction = 'horizontal', legend.position = 'top')
iris.gg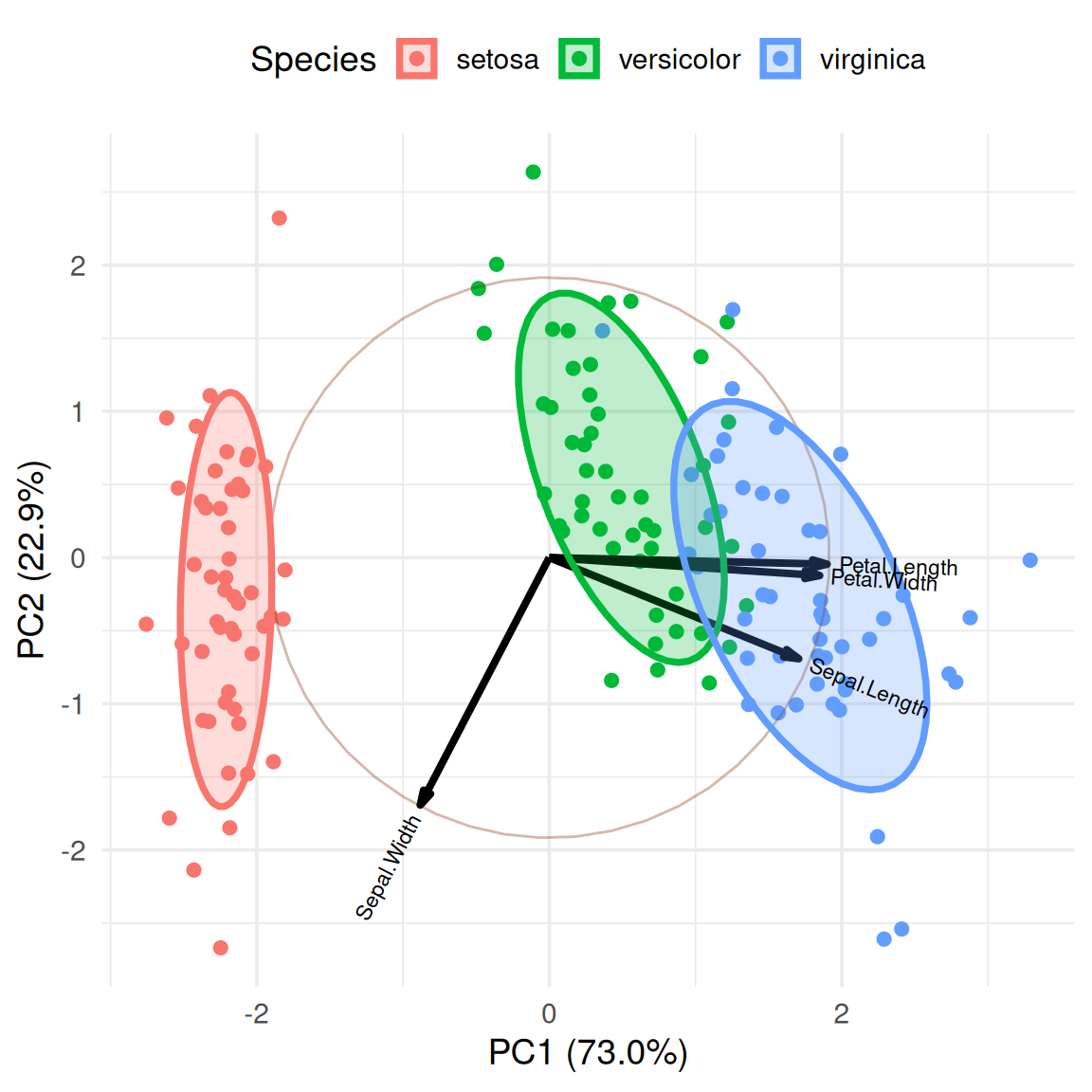
注:图题为基因名,横轴为PC1,纵轴为PC2,图中箭头所示向量长短代表差异贡献,方向为与主成分的相关性,颜色为不同种类的鸢尾花。
我们可以发现PC1和PC2可将三组样品明显分开,可以看到向量Petal.Length,Petal.width与X轴近平行,表示PC1的差异主要由此变量贡献,而PC2的差异则主要由Sepal.Width贡献。每个向量之间夹角大于90°代表负相关,夹角小于90%的代表正相关。
2. 增加聚类标签
我们可以通过计算聚类中心,添加标签,更直观地展示不同类别地名称。
group.labs <-
iris.gg$data %>%
dplyr::summarise(xvar = mean(xvar),
yvar = mean(yvar), .by = groups)
group.labs groups xvar yvar
1 setosa -2.2099215 -0.2870013
2 versicolor 0.4931384 0.5465027
3 virginica 1.7167831 -0.2595014iris.gg + geom_label(data = group.labs,
aes(x = xvar, y=yvar, label=groups),
size = 5) +
theme(legend.position = "none")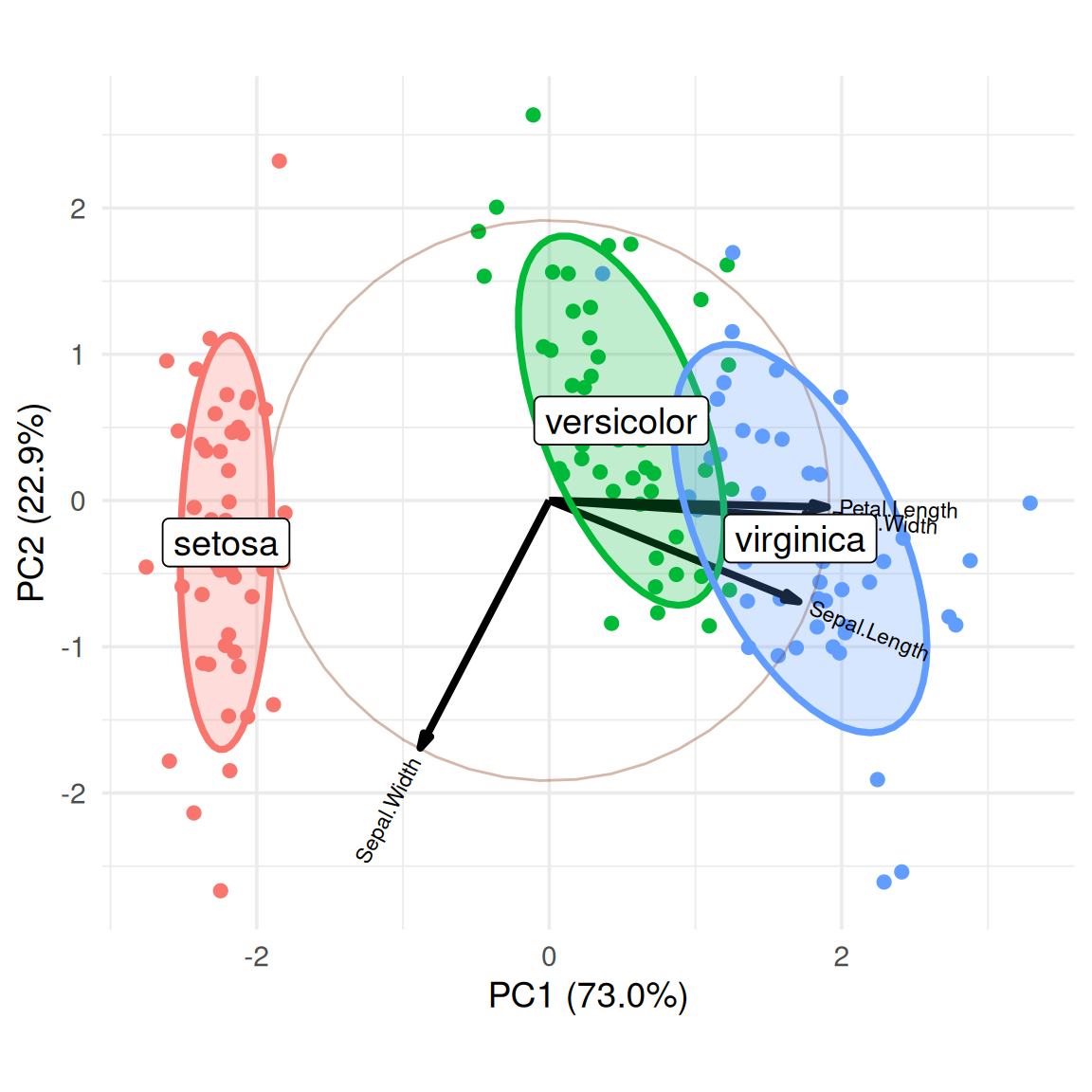
3. 使用样本名替代点
为了展示不同样本在PCA分析中所处地位置,我们可以通过设置labels来进行展示。
ggbiplot(iris.pca, obs.scale = 1,
var.scale = 1,
groups = iris$Species,
labels = rownames(iris),
point.size=2,
varname.size = 3,
varname.color = "black",
varname.adjust = 1.2,
ellipse = TRUE,
circle = TRUE) +
labs(fill = "Species", color = "Species") +
theme_minimal(base_size = 14) +
theme(legend.direction = 'horizontal', legend.position = 'top')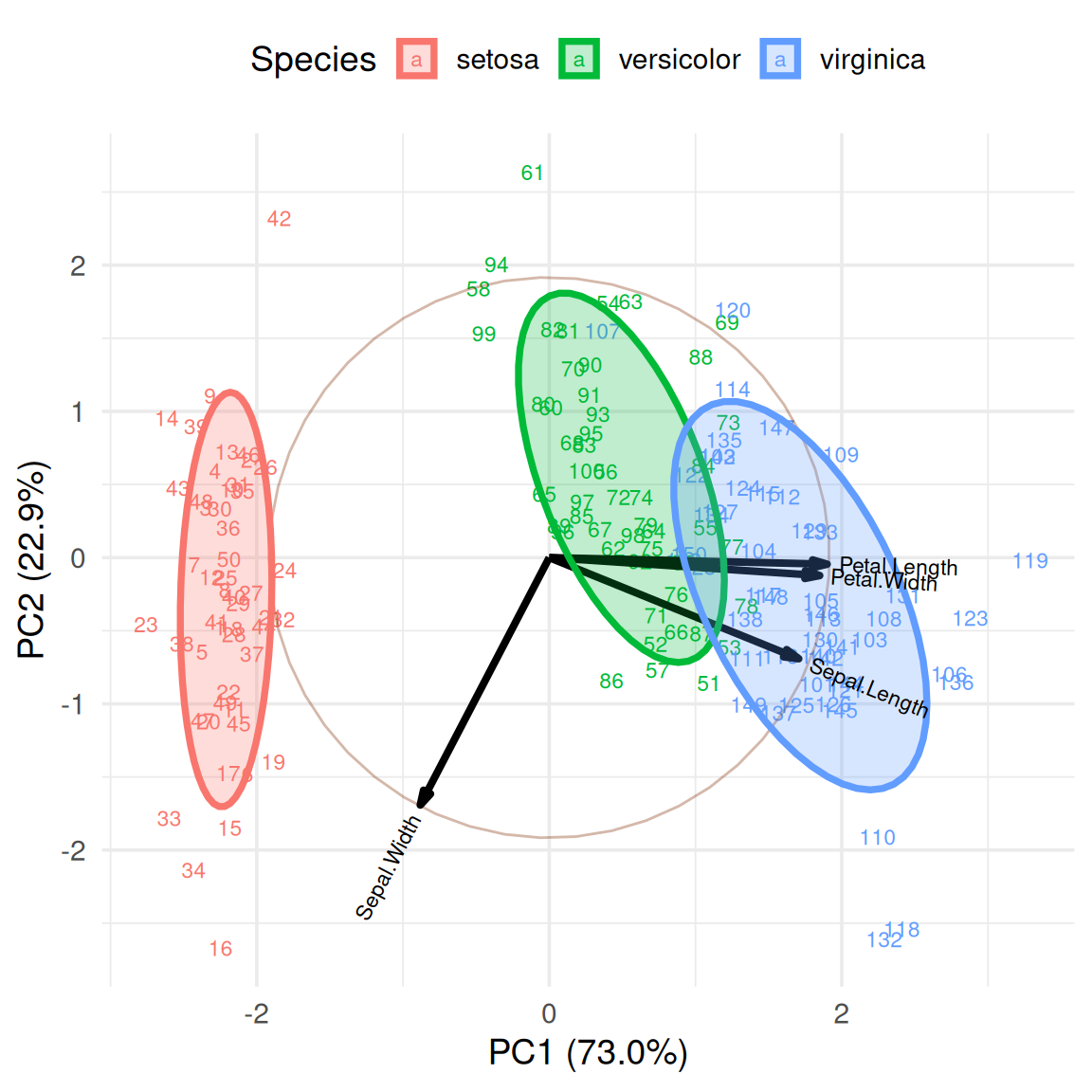
应用场景
1. 宏基因组主成分分析载荷图
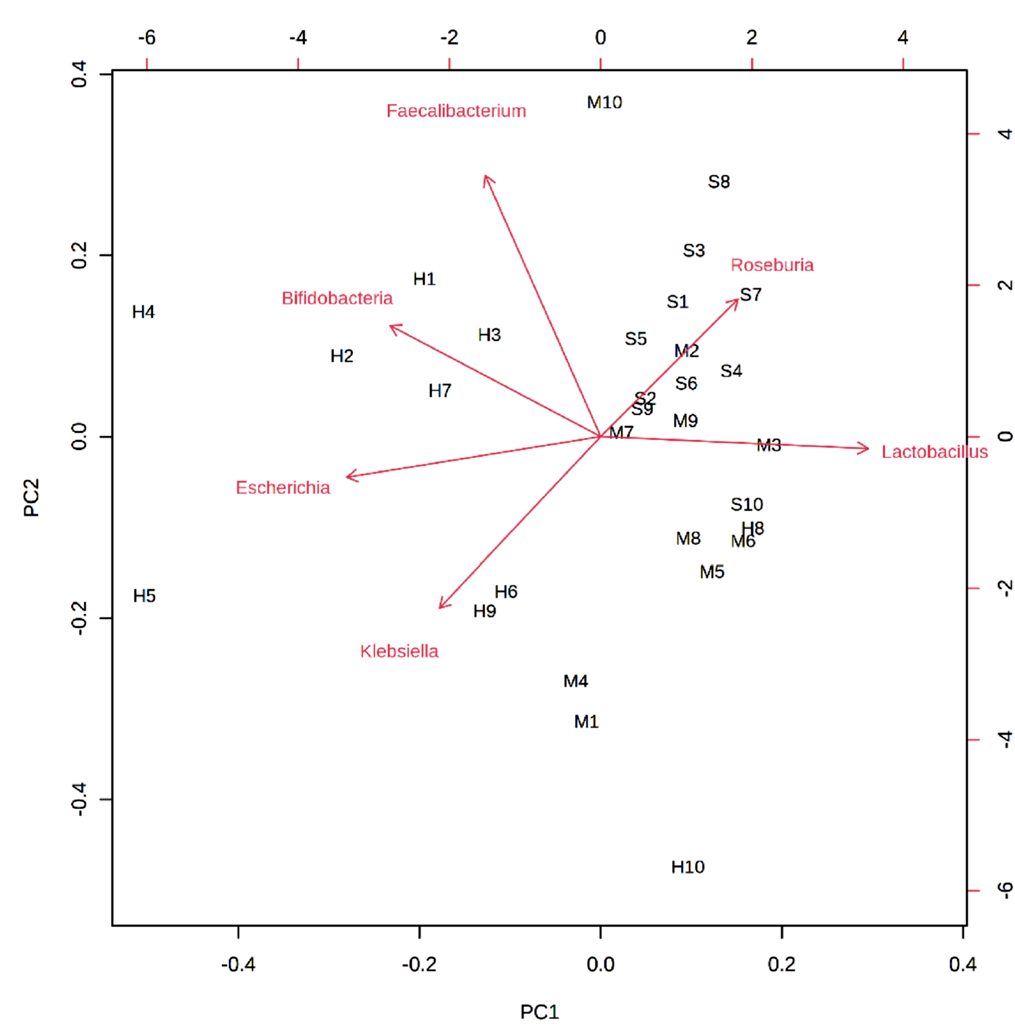
箭头代表菌种,字母代表样本,菌种与样本地距离越近表明相关性越高,图中与H1,H2,H3,H4,H7菌种最近的菌种是双歧杆菌属(bifidobacteria),表明该菌种是对照组中的优势菌种。[1]
2. 转录组主成分分析载荷图
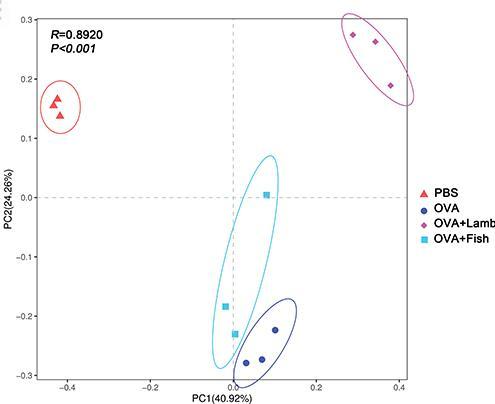
四组之间有显著差异,其中PC1和PC2的贡献值分别为40.92%和24.26%,PC2中,对照组(PBS)与哮喘+羊肉组(OVA+Lamb)正向分布,哮喘+鱼肉组(OVA+fish)与哮喘组(OVA)主要呈负向分布,对照组与其余三组有显著差异,哮喘+羊肉组(OVA+Lamb)与哮喘+鱼肉组(OVA+Fish)有显著分离。[2]
参考文献
[1] Balasubramaniam C, Mallappa RH, Singh DK, et al. Gut bacterial profile in Indian children of varying nutritional status: a comparative pilot study. Eur J Nutr. 2021;60(7):3971-3985. doi:10.1007/s00394-021-02571-7.
[2] Zheng HC, Wang YA, Liu ZR, et al. Consumption of Lamb Meat or Basa Fish Shapes the Gut Microbiota and Aggravates Pulmonary Inflammation in Asthmatic Mice. J Asthma Allergy. 2020;13:509-520. Published 2020 Oct 19. doi:10.2147/JAA.S266584.
