# 安装包
if (!requireNamespace("readr", quietly = TRUE)) {
install.packages("readr")
}
if (!requireNamespace("rms", quietly = TRUE)) {
install.packages("rms")
}
if (!requireNamespace("survival", quietly = TRUE)) {
install.packages("survival")
}
if (!requireNamespace("regplot", quietly = TRUE)) {
install.packages("regplot")
}
# 加载包
library(readr)
library(rms)
library(survival)
library(regplot)诺莫图
诺莫图(Nomogram),简单来说,就是将Logistic回归或Cox回归的结果通过图形化方式展示出来。它根据每个自变量的回归系数来制定评分标准,给每个自变量的取值分配一个分数。然后,针对每个患者,计算出一个总分,并通过转换函数,将得分转化为该患者发生特定结局的概率。
常见的列线图的名称主要包括三个部分: 1. 预测模型中的变量名称:例如图中的“stage”(阶段)、“gender”(性别)、“age”(年龄)等,每个变量对应的线段上标有刻度,表示该变量的取值范围,线段的长度反映该因素对结局事件的影响程度。 2. 得分:包括单项得分(图中的“β(X-m) terms”)和总得分(“Total score”)。单项得分表示每个变量在不同取值下的分数,总得分则是所有变量分数的加和。 3. 预测概率:例如“mean status”(平均状态),表示死亡的概率。
示例
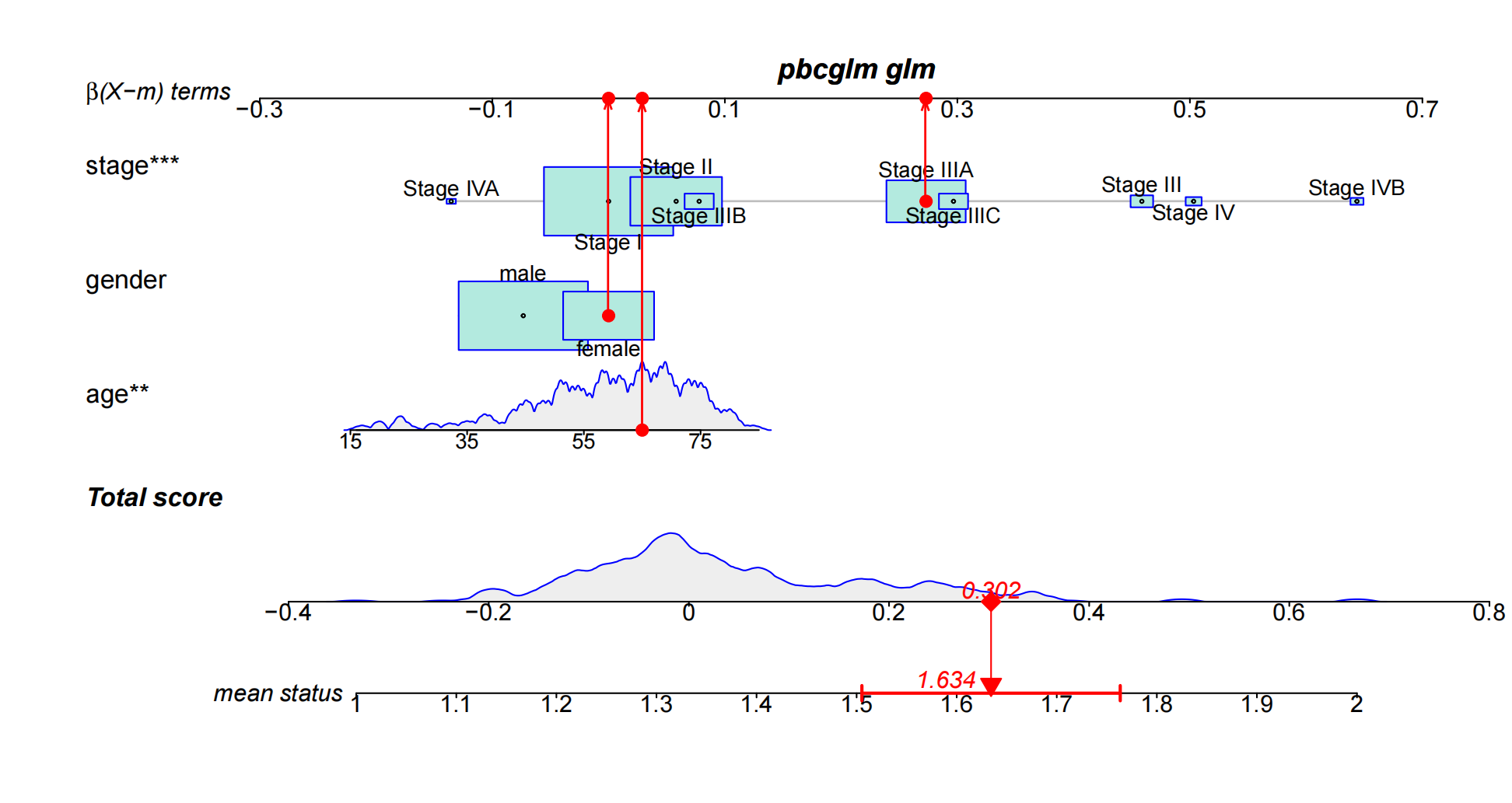
如红色箭头所示,找出该患者每个变量对应的单项得分。最后将所有变量的单项得分相加,得到患者的总得分,并以总得分为基础,再向下画一条垂直线,就可以知道该患者侵袭性肺腺癌的患病风险。
环境配置
系统要求: 跨平台(Linux/MacOS/Windows)
编程语言:R
依赖包:
readr,rms,survival,regplot
sessioninfo::session_info("attached")─ Session info ───────────────────────────────────────────────────────────────
setting value
version R version 4.5.2 (2025-10-31)
os Ubuntu 24.04.3 LTS
system x86_64, linux-gnu
ui X11
language (EN)
collate C.UTF-8
ctype C.UTF-8
tz UTC
date 2026-02-02
pandoc 3.1.3 @ /usr/bin/ (via rmarkdown)
quarto 1.8.27 @ /usr/local/bin/quarto
─ Packages ───────────────────────────────────────────────────────────────────
package * version date (UTC) lib source
Hmisc * 5.2-5 2026-01-09 [1] RSPM
readr * 2.1.6 2025-11-14 [1] RSPM
regplot * 1.1 2020-06-26 [1] RSPM
rms * 8.1-0 2025-10-14 [1] RSPM
survival * 3.8-3 2024-12-17 [3] CRAN (R 4.5.2)
[1] /home/runner/work/_temp/Library
[2] /opt/R/4.5.2/lib/R/site-library
[3] /opt/R/4.5.2/lib/R/library
* ── Packages attached to the search path.
──────────────────────────────────────────────────────────────────────────────数据准备
数据下载于Xena浏览器 [2] 中GDC TCGA Liver Cancer (LIHC)数据库的Phenotype文件。
## 载入数据
clinical <- readr::read_tsv("https://bizard-1301043367.cos.ap-guangzhou.myqcloud.com/TCGA-LIHC.clinical.tsv")
LIHC <- cbind(clinical$sample,clinical[,c('gender.demographic',
'vital_status.demographic',
'days_to_death.demographic',
'age_at_index.demographic',
'ajcc_pathologic_stage.diagnoses')])
colnames(LIHC) <- c('bcr_patient_barcode','gender','status','time','age','stage')
table(LIHC$status)
Alive Dead Not Reported
265 172 2 LIHC <- LIHC[LIHC$status != 'Not Reported',]
LIHC$status <- as.numeric(ifelse(LIHC$status=='Dead','2','1') ) # 在nomogram中死亡为2可视化
1. 基础诺莫图
基础诺莫图显示了使用广义线性模型对生存状态(status)与年龄(age)、性别(gender)、疾病阶段(stage)建模的结果。
# 基础诺莫图
dd=datadist(LIHC)
options(datadist="dd")
## 构建logist模型,绘制诺莫图
f1 <- lrm(status ~ age + gender + stage , data = LIHC)
nom <- nomogram(f1, fun=plogis, lp=F, funlabel="Risk")
plot(nom)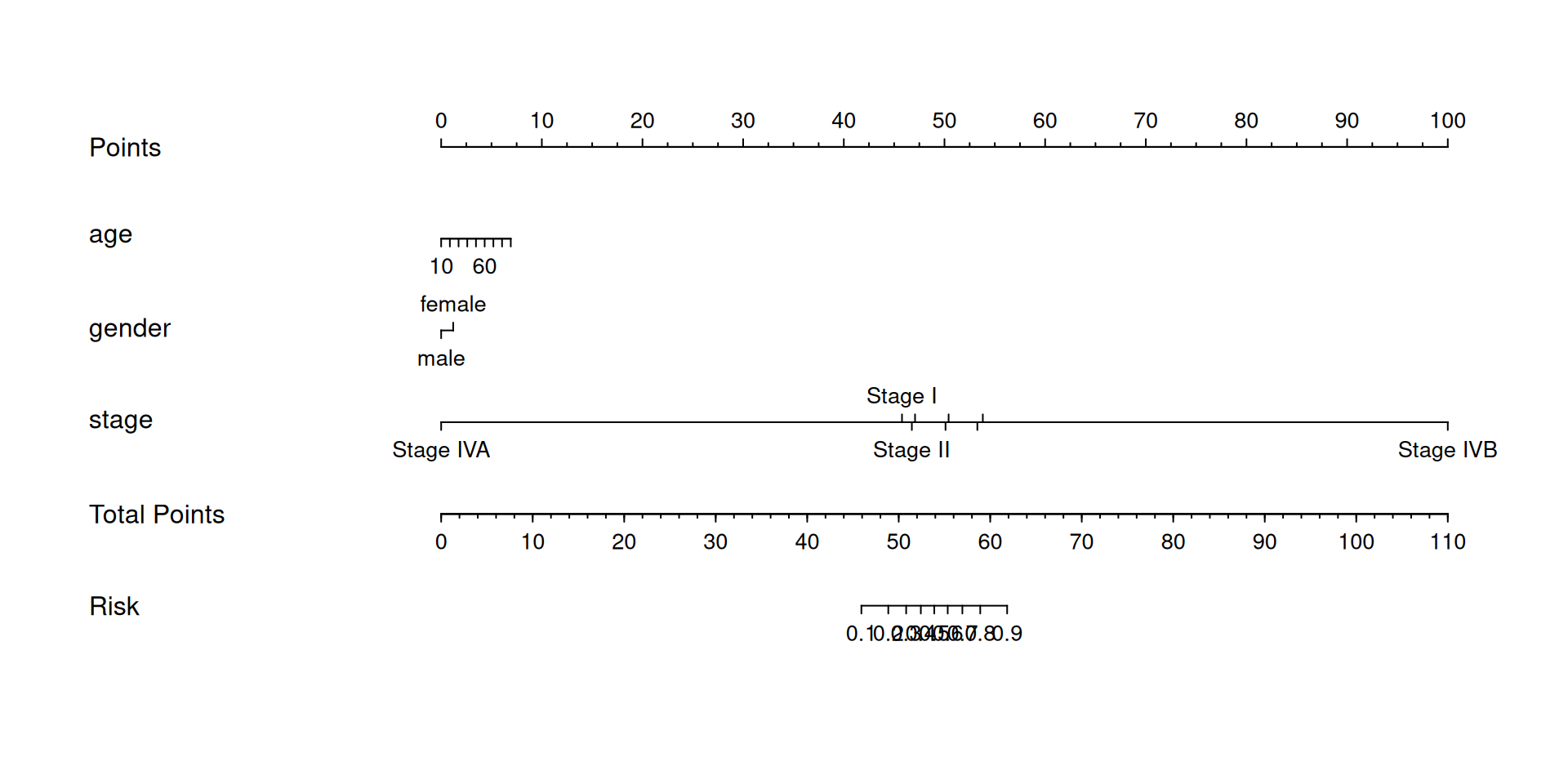
我们也可以使用诺莫图展示cox回归模型对生存时间(time)、生存状态(status)与年龄(age)、性别(gender)、疾病阶段(stage)建模的结果。
## 构建COX比例风险模型
f2 <- psm(Surv(time,status) ~ age+gender+stage,data = LIHC, dist='lognormal')
med <- Quantile(f2) # 计算中位生存时间
surv <- Survival(f2) # 构建生存概率函数
## 绘制COX回归中位生存时间的Nomogram图
nom <- nomogram(f2, fun=function(x) med(lp=x),funlabel="Median Survival Time")
plot(nom)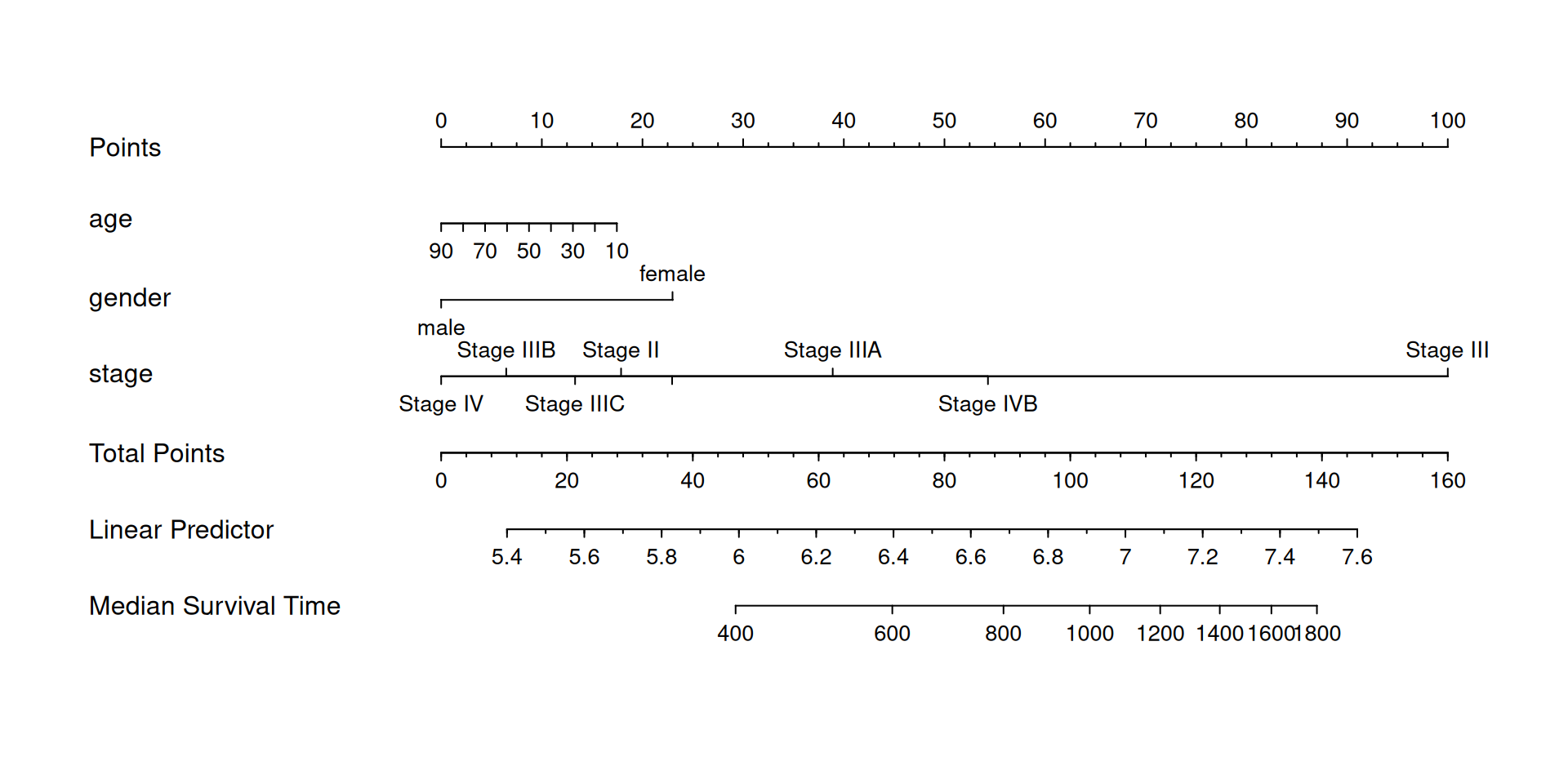
我们还可以使用诺莫图对预测的3年-5年-生存概率进行绘制。
## LIHC数据的time是以”天“为单位,此处绘制3年,5年的生存概率
nom <- nomogram(f2, fun=list(function(x) surv(1095, x),
function(x) surv(1825, x),
function(x) med(lp=x)),
funlabel=c("3-year Survival Probability", "5-year Survival Probability","Median Survival Time"))
plot(nom, xfrac=.2)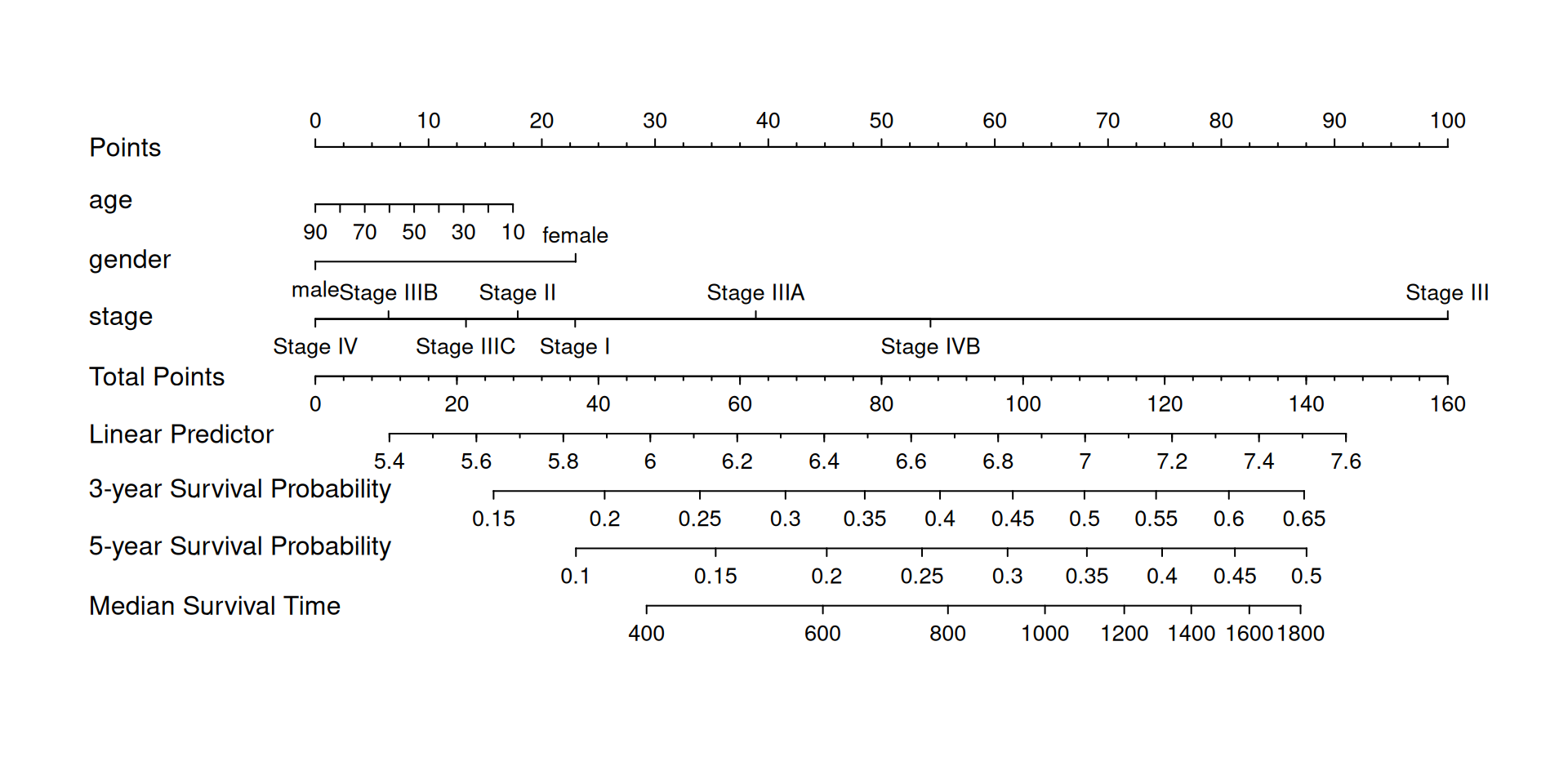
2. 美化诺莫图
除了使用nomogram进行建模绘图外,我们还可以使用survival包和regplot包配合进行建模与绘图,下图是使用广义线性模型进行模型的构建与绘制。
# 展示逻辑回归,支持"lm", "glm", "coxph", "survreg" "negbin"
## 绘制逻辑回归图,显示优势尺度和置信区间
lihcglm <- glm(status ~ age + gender + stage, data=LIHC )
regplot(lihcglm,
observation=LIHC[1,],
odds=TRUE,
interval="confidence")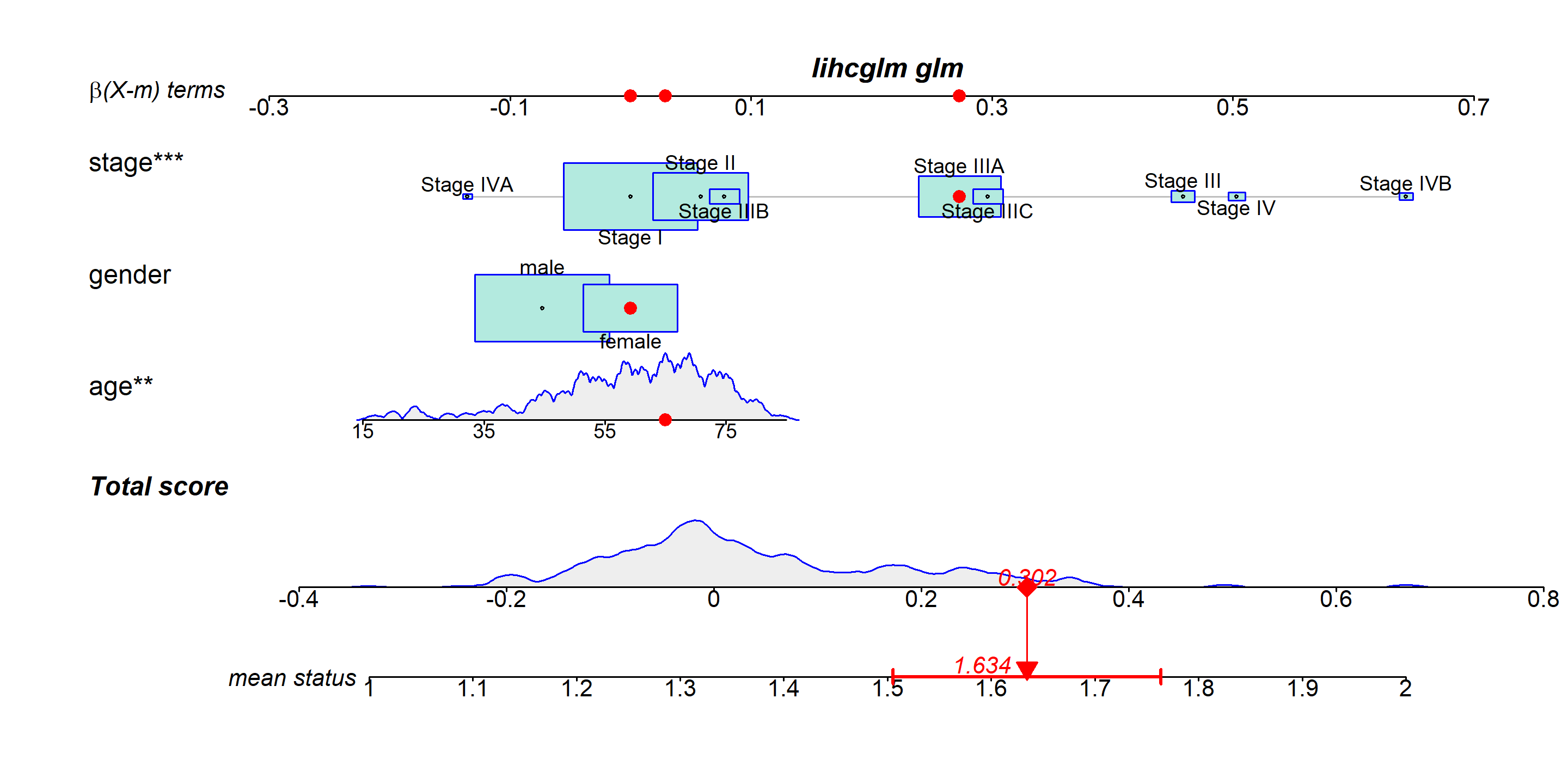
下图是使用广义线性模型进行模型的构建与绘制。
lihccox <- coxph(formula = Surv(time,status) ~ age+gender+stage , data = LIHC)
regplot(lihccox,
observation=LIHC[2,], #对观测2的指标在列线图上进行计分展示
failtime = c(1095,1825),#预测3年和5年的死亡风险,此处单位是day
prfail = TRUE, #cox回归中需要TRUE
showP = T, #是否展示统计学差异
droplines = F,#观测2示例计分是否画线
interval="confidence") #展示观测的可信区间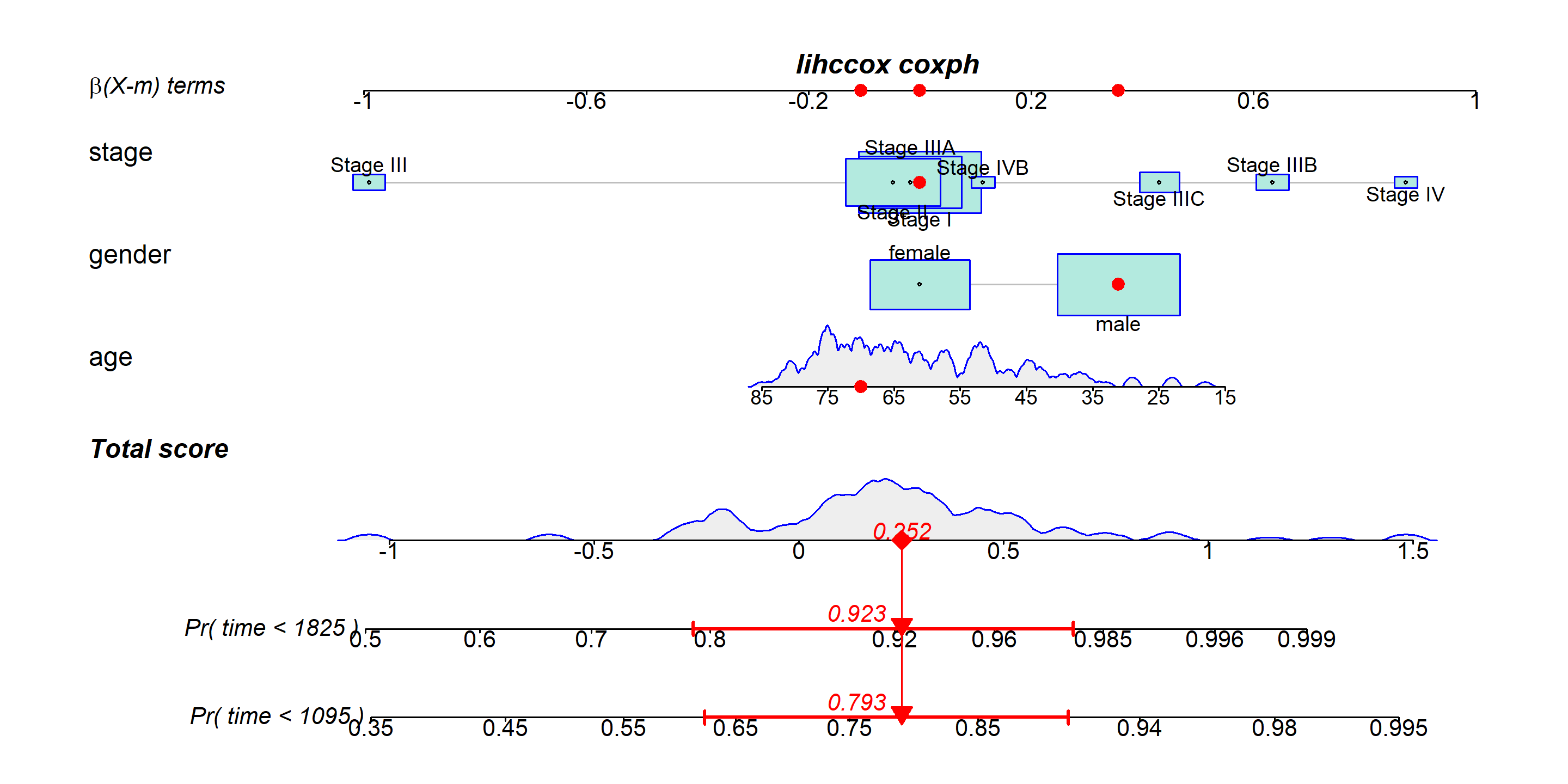
应用场景
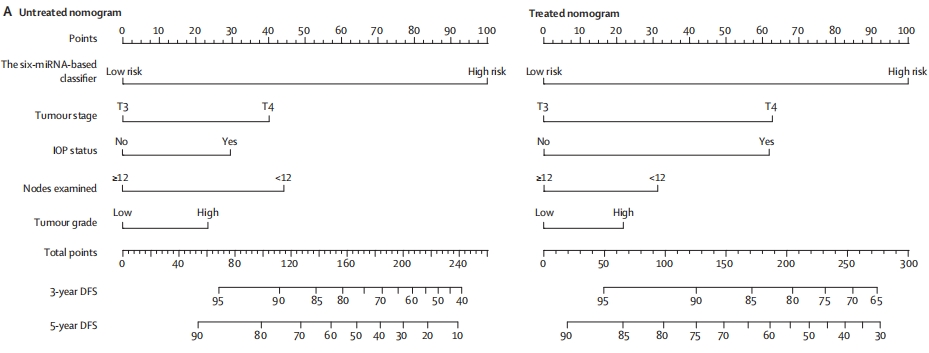
该图为两个用于临床应用的示意图,整合了基于 6miRNA 的分类器和四个临床病理风险因素,以预测哪些患者可能从 II 期结肠癌手术后的辅助化疗中受益。[2]
参考文献
[1] Goldman MJ, Craft B, Hastie M, et al. Visualizing and interpreting cancer genomics data via the Xena platform. Nat Biotechnol. 2020;38(6):675-678. doi:10.1038/s41587-020-0546-8
[2] Zhang JX, Song W, Chen ZH, et al. Prognostic and predictive value of a microRNA signature in stage II colon cancer: a microRNA expression analysis [published correction appears in Lancet Oncol. 2014 Jan;15(1):e4]. Lancet Oncol. 2013;14(13):1295-1306. doi:10.1016/S1470-2045(13)70491-1
