生信爱好者周刊(第 94 期):非线性的世界,线性的你¶
这里记录每周值得分享的生信相关内容,周日发布。
本杂志开源(GitHub: openbiox/weekly),欢迎提交 issue,投稿或推荐生信相关内容。
封面图¶
 Despite the wind and rain, I am still standing still-任凭风吹雨打 我自巍然不动
Despite the wind and rain, I am still standing still-任凭风吹雨打 我自巍然不动
本周话题:非线性的世界,线性的你¶
现实世界是非线性的、非因果的,而人的思维是线性的、因果关系的,会不知不觉从单一的线性因素,去解读非线性关系。 你不断加大努力的程度,长时间工作、长期加班、没有个人生活,会带来成功吗?很可能不会,因为两者之间没有因果关系。
@kktjmac 因为这个非线性的世界,所以更应该从多元化、全局化的角度去认识、理解生活与工作。
生信研究¶
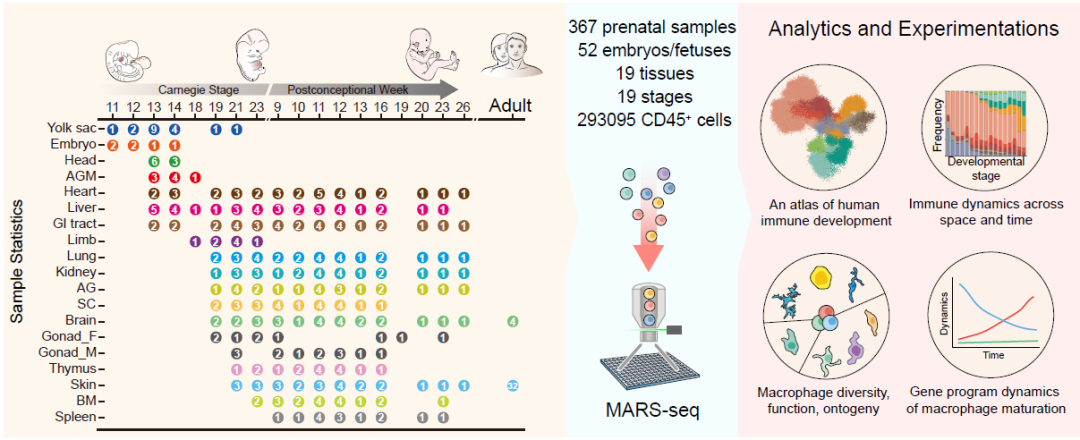
中国科学院深圳先进技术研究院李汉杰课题组联合深圳市宝安区妇幼保健院、深圳大学、上海交通大学和复旦大学等单位在Cell期刊上发表了题为“An immune cell atlas reveals the dynamics of human macrophage specification during prenatal development”的文章。研究团队结合单细胞转录组测序、先进的生物信息学手段、多重免疫荧光染色、体外功能实验等技术构建了横跨18个发育阶段、19种组织的人类胚胎免疫系统发育高分辨率图谱。研究团队重点关注了最具组织特异性的巨噬细胞,鉴定了15种巨噬细胞亚型,其中包括两种新的巨噬细胞亚型,即存在于表皮、睾丸、心脏等外周组织的类小胶质细胞(Microglia-like cells)和具有血管生成促进功能并广泛分布于多个组织中的促血管生成巨噬细胞(Proangiogenic macrophages, PraM)。该研究通过构建人类产前免疫细胞发育的时空动态图谱,揭示了多种巨噬细胞亚型在发育过程中的分化起源、空间定位、功能特征及转录调控机制。
- 可视化平台链接:http://119.8.233.169/
- 论文链接:https://doi.org/10.1016/j.cell.2023.08.019
2、Nature Methods | 单分子测序平台的scNanoHi-C技术,可精准检测单细胞高阶染色质互作
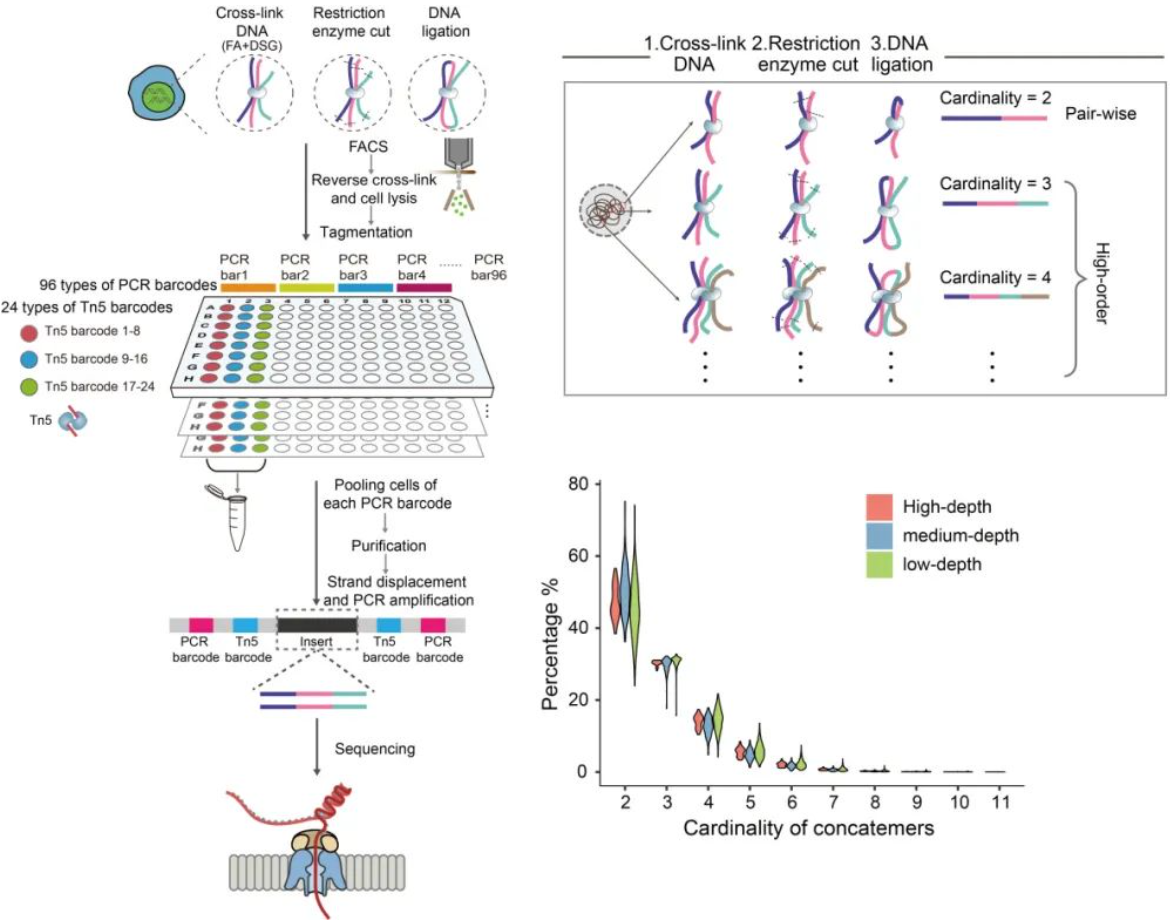
这是一篇汤富酬课题组在Nature Methods上发表的文章,该研究在国际上率先使用单分子测序平台开发了一种基于邻近连接的单细胞染色质构象捕获方法,称为scNanoHi-C。该方法实现了在单细胞水平的高阶染色质相互作用检测,并且在通量上具有很好的灵活性,能够满足不同的实验需求。
- 论文链接:https://www.nature.com/articles/s41592-023-01978-w
3、Nature Biotechnology | 全球首套多组学标准物质“中华家系1号”最新成果
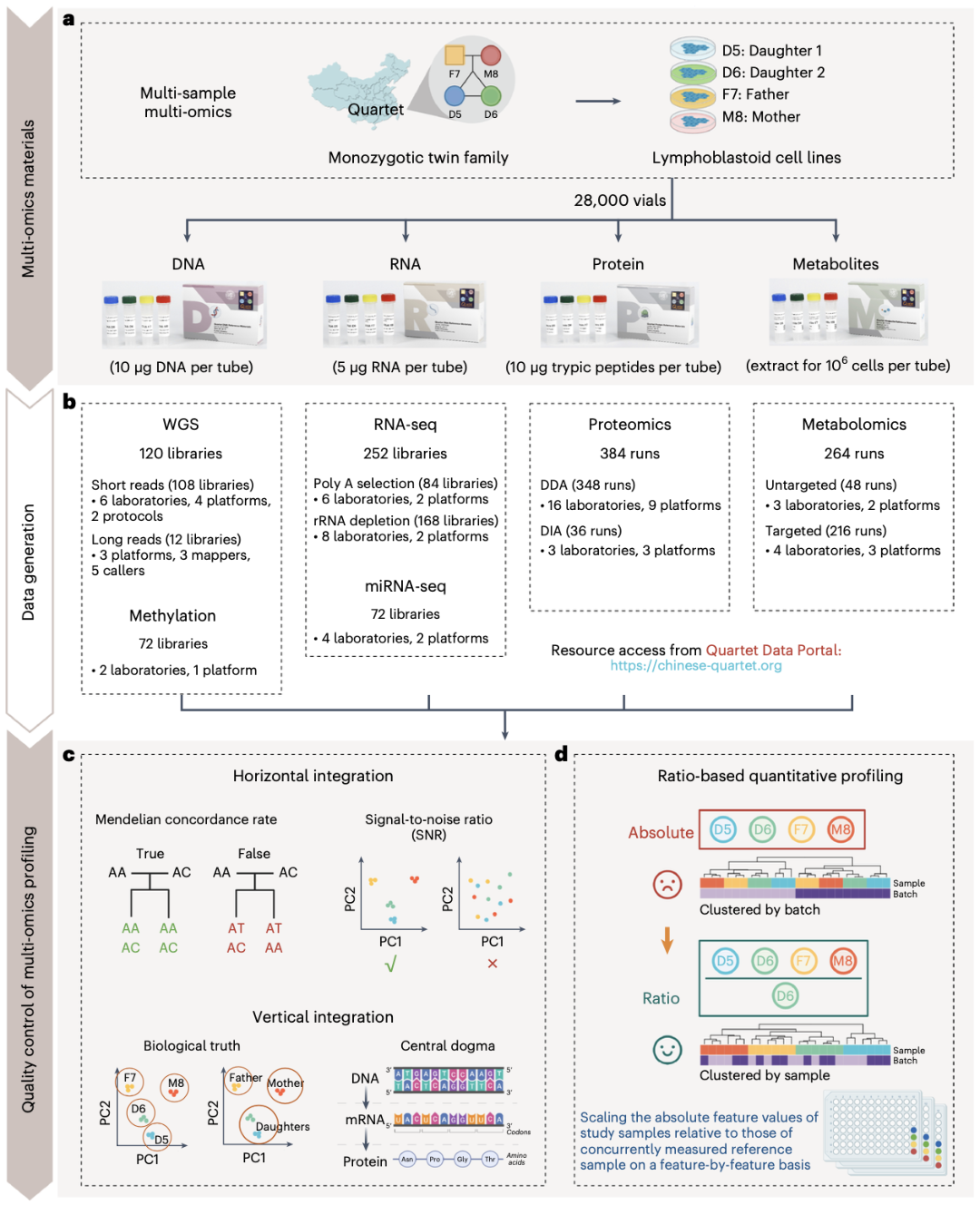
国际学术期刊《自然·生物技术》(Nature Biotechnology)在线发表了由复旦大学/上海国际人类表型组研究院石乐明、郑媛婷团队联合中国计量科学研究院方向、董莲华团队,国家卫健委临床检验中心李金明、张瑞团队共同研发的全球首套多组学标准物质“中华家系1号”的最新研究成果。同期刊发的2篇科研论文分别聚焦:“使用基于中华家系1号标准物质的相对定量进行多组学数据整合(Multi-omics data integration using ratio-based quantitative profiling with Quartet reference materials)”和“中华家系1号 RNA标准物质与基于比值的分析方法提高了转录组数据的质量(Quartet RNA reference materials and ratio-based profiling for assessing and improving the quality of transcriptomic data)”。这也标志着中国科学界自主研制、获批为“国家一级标准物质”的“中华家系1号(Quartet)”多组学标准物质的研发和效用得到了国际同行的认可,开创了生物医学“度量衡”新体系,将提升生命科学创新的源头质量,为全球推进人类表型组计划奠定坚实的标准基础。
- 论文链接:https://www.nature.com/articles/s41587-023-01934-1
- 论文链接:https://www.nature.com/articles/s41587-023-01867-9
博文资讯¶
4、视频:研究蛋白质与DNA相互作用的新技术–CUT&Tag
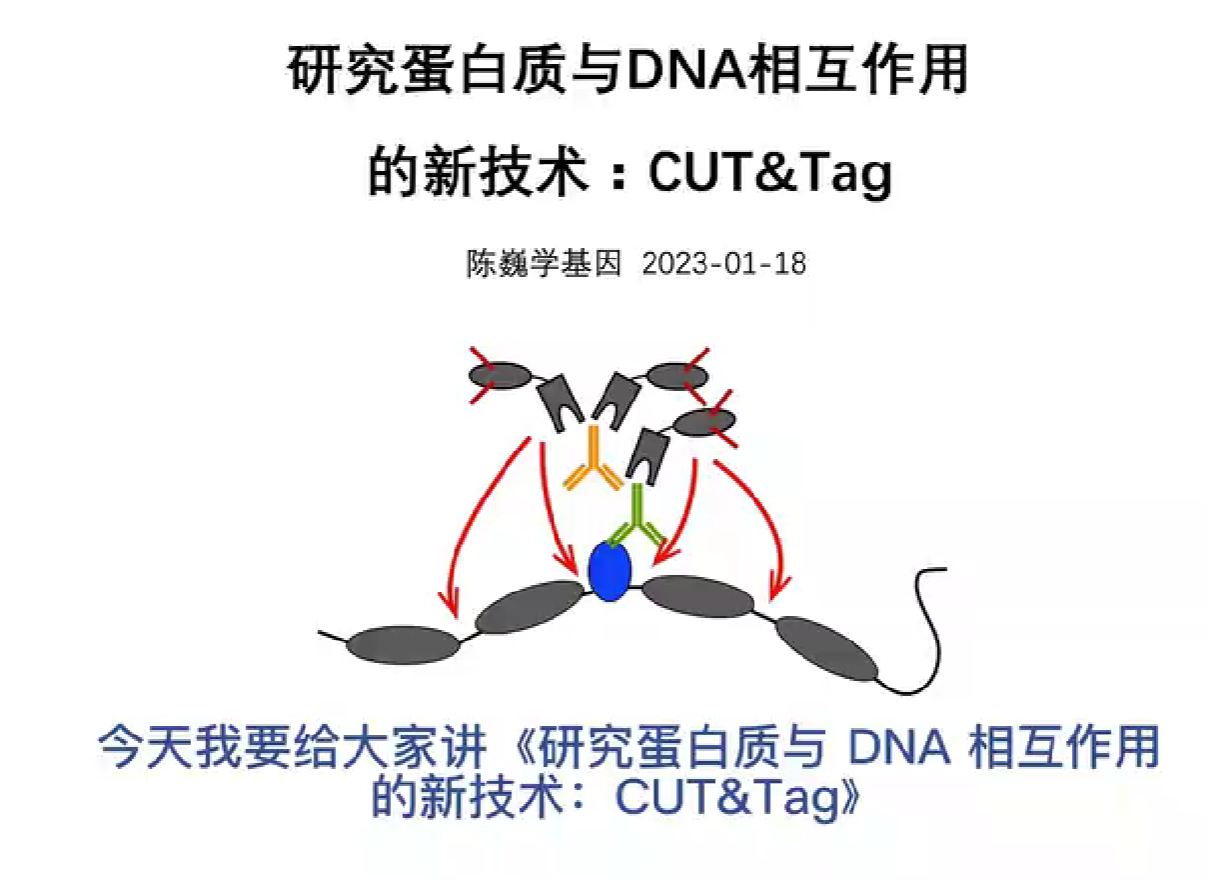
CUT&Tag 是一种新的检测目标蛋白质与染色质 DNA 相互作用的一种技术。CUT&Tag 方法比经典的 ChIP-seq 方法的信噪比提升了10倍以上,并且所需的起始样本细胞的数量少了10倍,实验操作时间只需 1~2 天。 它的核心技术点在于,利用抗体把目标蛋白和基因工程改造过的 Tn5 转座酶吸附在一起,利用 Tn5 酶的转座功能对目标蛋白吸附区附近的 DNA 进行切断,并在切下的片段两端加上标签序列,然后构建成深度测序文库。
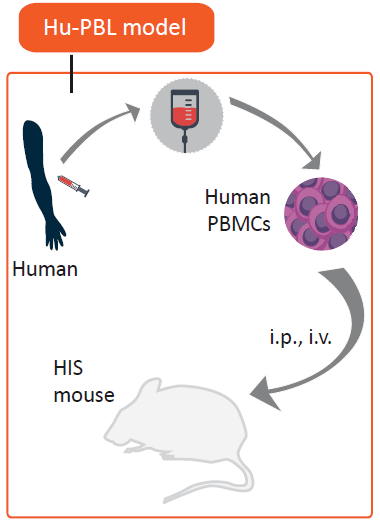
一篇介绍了免疫系统人源化小鼠模型,以及它在人类疾病和生理研究中的应用的文章。免疫系统人源化小鼠模型通过将人类的造血细胞或组织植入免疫缺陷小鼠体内,从而建立起近似于人类的免疫系统。目的是弥补普通小鼠模型与人类在免疫系统上的差异。文章根据重建免疫系统的不同方法,将人源化小鼠模型分为三类:Hu-PBMC模型、Hu-HSC模型和Hu-BLT模型。它们各有优劣分别适用于不同研究领域。
6、IGV数据可视化

IGV(Integrative Genomics Viewer)是一种开源基因组浏览器,是一种可视化工具,用于观察基因组数据集中具有生物学意义的模式,包括序列数据,基因模型,比对和DNA微阵列数据。本文对 IGV 的界面和操作进行了基本的介绍。
工具¶
7、SCENICprotocol | 可扩展的单细胞基因调控网络分析流程
SCENICprotocol介绍了如何使用pySCENIC对单细胞数据进行基因调控网络推断分析,并结合既往的“最佳实践”分析流程进行分析。具体包括: - 交互式分析的Jupyter notebooks - Nextflow DSL1工作流程,提供半自动化和简化的方法来运行这些步骤 - 关于pySCENIC的安装、使用和下游分析的详细信息
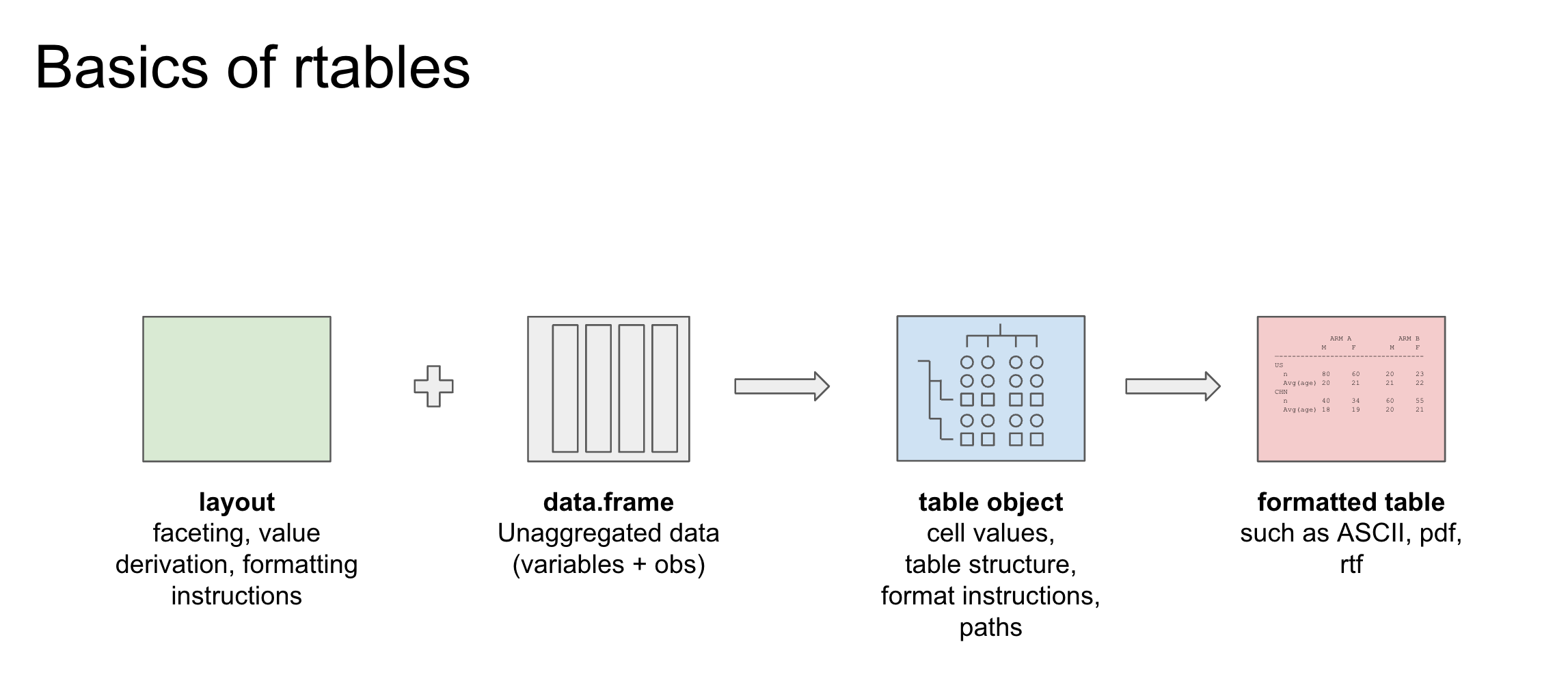
rtables包可以通过R创建、展示复杂的表格。rtable中的单元格可以包含任何高维数据结构,同时支持输出ascii html、pdf以及Power Point等多种格式(通过转换为flextable对象)。

这个GitHub库提供了一个单细胞RNA测序数据分析流程。本分析流程使用Docker容器技术打包相关分析软件,实现从原始数据预处理至下游分析的完整工作流。用户只需设置参数文件和提供数据,即可一键运行整个分析流程。该管道旨在简化和加速单细胞RNA测序数据分析,为研究人员提供一个分析平台,助力生物学发现。
- 工具链接:https://github.com/OpenGene/scrnapip
10、colorblindr | 针对色觉缺陷用户优化可视化结果的R包
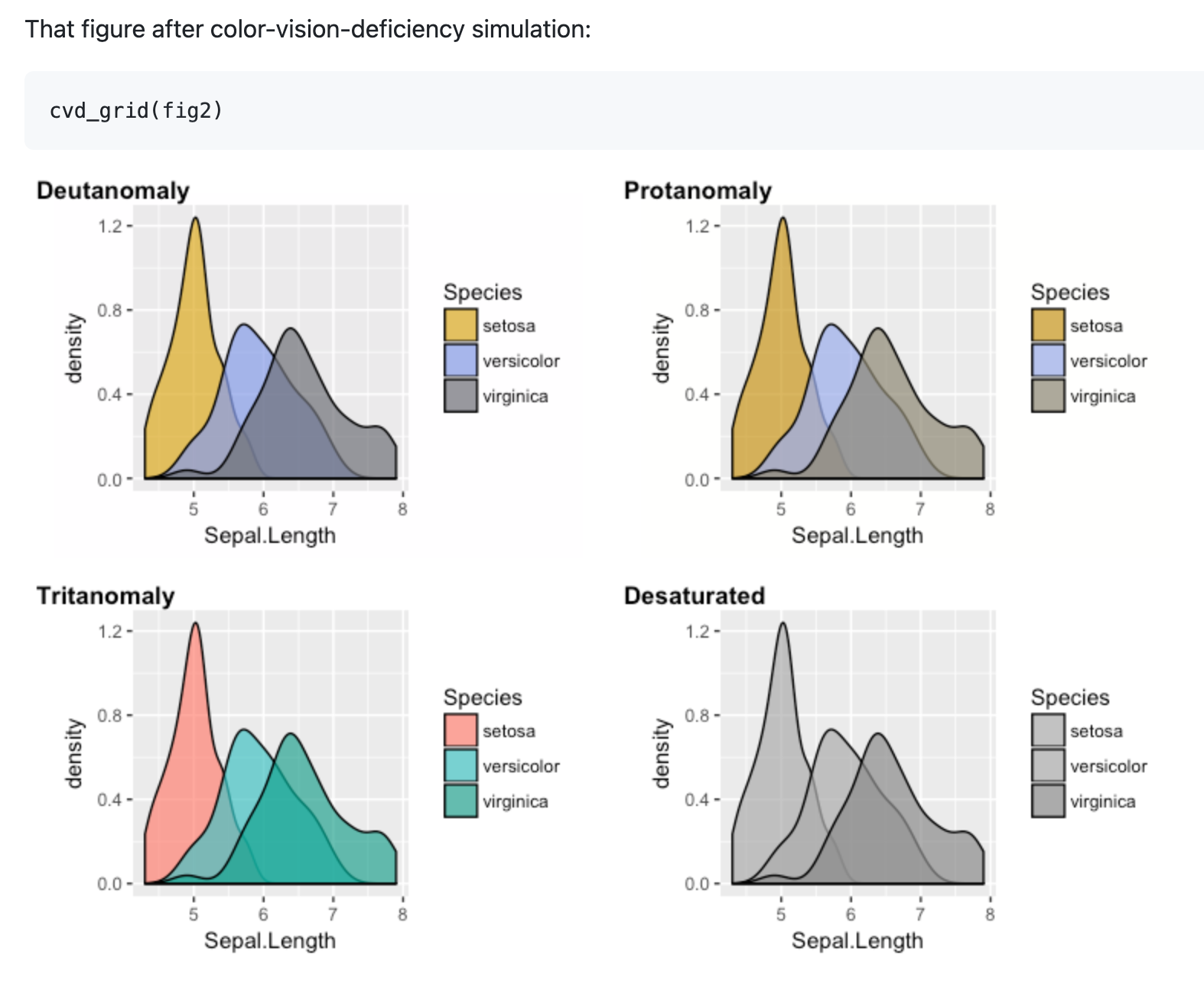
色觉缺陷用户可能对R中常规的配色方案无法辨识,针对这个问题,colorblindr包提供了相关解决方案。
资源¶
11、RM2Target | RNA修饰相关调控蛋白靶基因的综合数据库资源
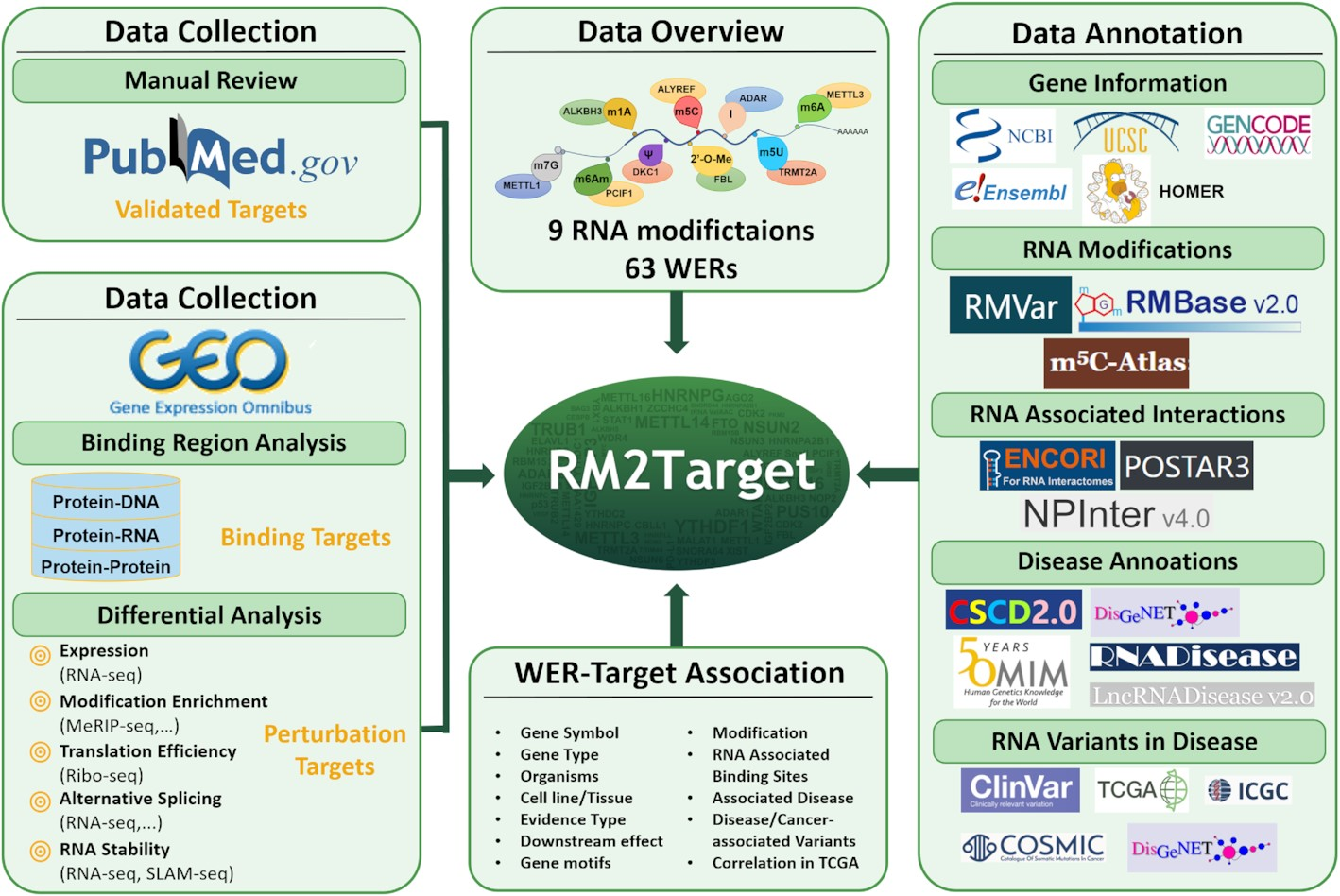
RM2Target收集了来自2个物种的419种不同的细胞系和组织的文献和公开数据集,涵盖了9种RNA修饰类型(m6A, m6Am, m5C, m5U, m1A, m7G, pseudouridine, 2’-O-Me, A-to-I)和63个相关的WER,目前共收录了1,801,980条记录,涉及1,619,653条WER-靶基因关联。
- 资源链接:http://rm2target.canceromics.org/#/home
- 论文链接:https://academic.oup.com/nar/article/51/D1/D269/6775386
12、RNA-seqblog | RNA测序技术和应用的博客网站
RNA-Seq Blog,一个关于RNA测序技术和应用的博客网站。它提供了以下内容:
- RNA-Seq的基本原理、优势和挑战
- RNA-Seq的最新进展和研究成果
- RNA-Seq的不同类型和方法,如单细胞RNA-Seq、转录组分析、拷贝数变异检测等
- RNA-Seq的相关工具、软件和数据库的介绍和评价
- RNA-Seq的相关课程、研讨会和招聘信息
历史上的本周¶
- 第54期:人类和人生的意义
贡献者(GitHub ID)¶
「Openbiox 生信周刊」运维小队:
@ShixiangWang(王诗翔)@kkjtmac(阚科佳)@NiEntropy(赵启祥)@He-Kai-fly(何凯)@JnanZhang(张佳楠)@Tomcxf(陈啸枫)@wangdepin(王德品)@kongjianyang(空间阳)
订阅¶
这个周刊每周日发布,同步更新在微信公众号「优雅R」(elegant-r)上。
微信搜索“优雅R”或者扫描二维码,即可订阅。

(完)