生信爱好者周刊(第 109 期):说说你今年用的顺手的工具吧?¶
这里记录每周值得分享的生信相关内容,周日发布。
本杂志开源(GitHub: openbiox/weekly),欢迎提交 issue,投稿或推荐生信相关内容。
封面图¶
 AI生成的巨型计算机横截面
来源:u/yoyoyodojo on Reddit
AI生成的巨型计算机横截面
来源:u/yoyoyodojo on Reddit
本周话题:说说你今年用的顺手的工具吧?¶
@kkjtmac 1.flomo浮墨笔记-记录碎片化的内容 2.ChatGPT Next Web-通过api使用ChatGPT 3.CodeRunner-Mac上的简易编程开发软件 4.无边记-苹果生态中的数字白板App,自由发挥整理各类资料 5.Notion-一款将笔记、知识库和任务管理整合的协作工具
生信研究¶
1、Nature | gnomAD数据库研究新成果:基于7.6万人WGS数据构建全基因组突变约束图谱
gnomAD是由美国Broad研究团队领导开发的,研究人员聚合并重新处理了gnomAD中的153,030个全基因组,将其与人类参考基因组GRCh38进行比对,保留了来自76,156个个体的全基因组数据,进一步开发了gnomAD(v3),构建了人类全基因组突变约束图谱(Gnocchi),以识别在突变时最有可能导致疾病的非编码区域。该研究为理解非编码区域的约束提供了新视角;Gnocchi图谱提高了人们对基因组功能遗传变异的识别及理解。
- 论文链接:https://doi.org/10.1038/s41586-023-06045-0
- 数据库链接:https://gnomad.broadinstitute.org/
2、Nature |全自动科研机器来了!4分钟设计整套复杂实验,自主完成实验全流程
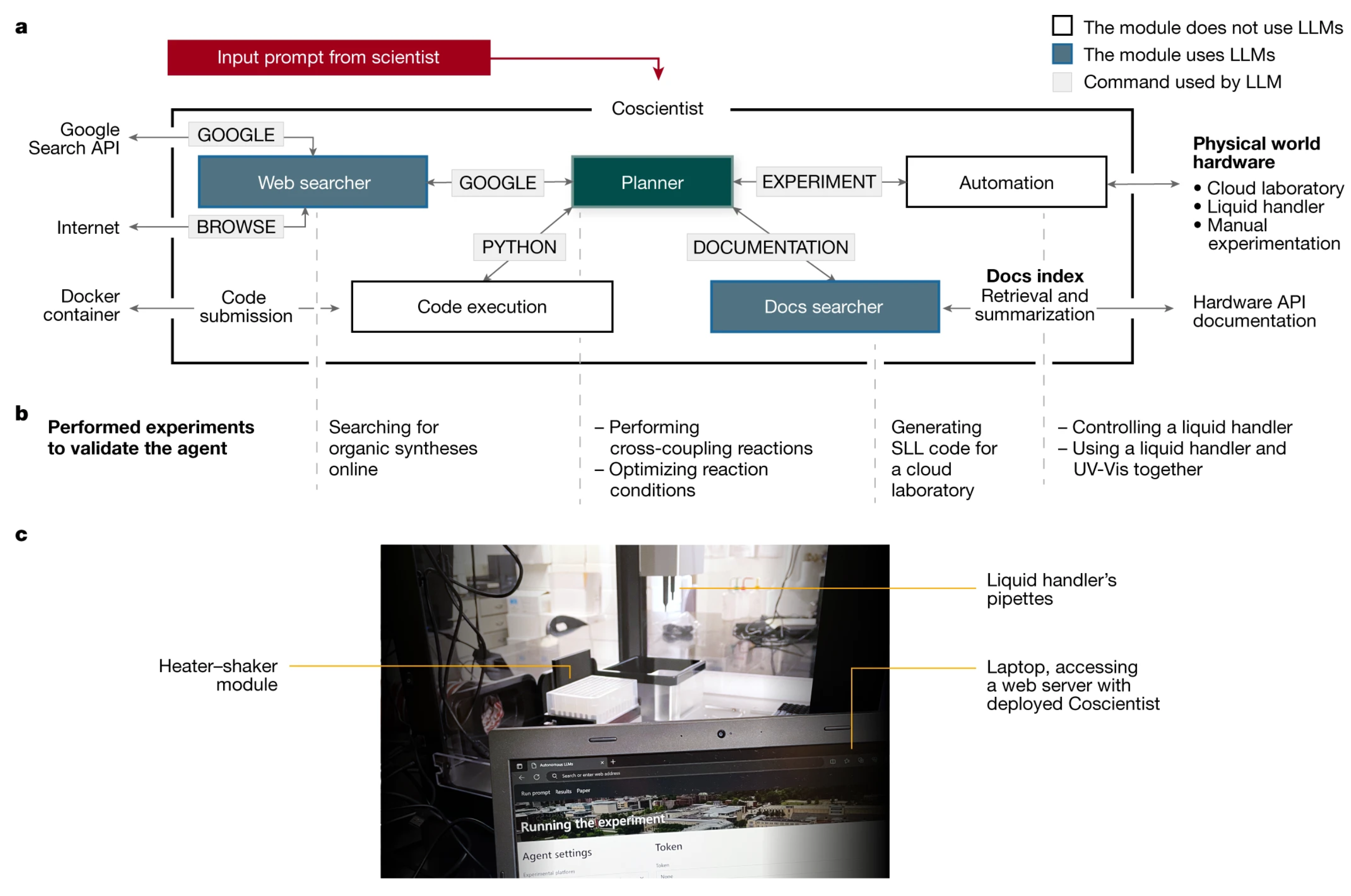
来自卡内基梅隆大学的研究团队开发了Coscientist——一款基于GPT-4等大型语言模型的AI系统,它可以快速、准确地自主完成从检索信息、规划及设计实验、编写程序、远程操控自动化系统来做实验再到分析数据的一整套流程。Coscientist的软件模块能够从互联网、文档数据和其他可用来源检索关于化合物的公开信息。通过学术期刊、维基百科、美国化学会等途径进行学习后,Coscientist能根据学到的信息指导自己的行动,进一步制定合成途径并决定实验方案。
- 论文链接:https://www.nature.com/articles/s41586-023-06792-0
3、Nature | 全方位绘制小鼠大脑图谱——约3200万个细胞,鉴别出约5300个细胞类型

2023年10-12月期间Nature上接连发布了10篇关于小鼠大脑图谱的文章,该系列文章由全球多个知名研究中心发表,利用多种技术包括scRNA-seq、snRNA-seq、snm3C-seq、MERFISH(Multiplexed error-robust fluorescence in situ hybridization)、Slide-seq、STARmap、SMART-seq、snmC-seq、snATAC-seq分析了小鼠大脑总计约3200万个细胞,鉴别出约5300个细胞类型,绘制了小鼠大脑详细的单细胞分子和空间图谱。
- 论文链接: https://doi.org/10.1038/s41586-023-06824-9 https://doi.org/10.1038/s41586-023-06819-6 https://doi.org/10.1038/s41586-023-06805-y https://doi.org/10.1038/s41586-023-06823-w https://doi.org/10.1038/s41586-023-06808-9 https://doi.org/10.1038/s41586-023-06818-7 https://doi.org/10.1038/s41586-023-06812-z https://doi.org/10.1038/s41586-023-06817-8 https://doi.org/10.1038/s41586-023-06638-9 https://doi.org/10.1038/s41586-023-06569-5
4、Nature Genetics | 基于健康单细胞参考图谱准确识别scRNA-seq数据中疾病相关细胞状态
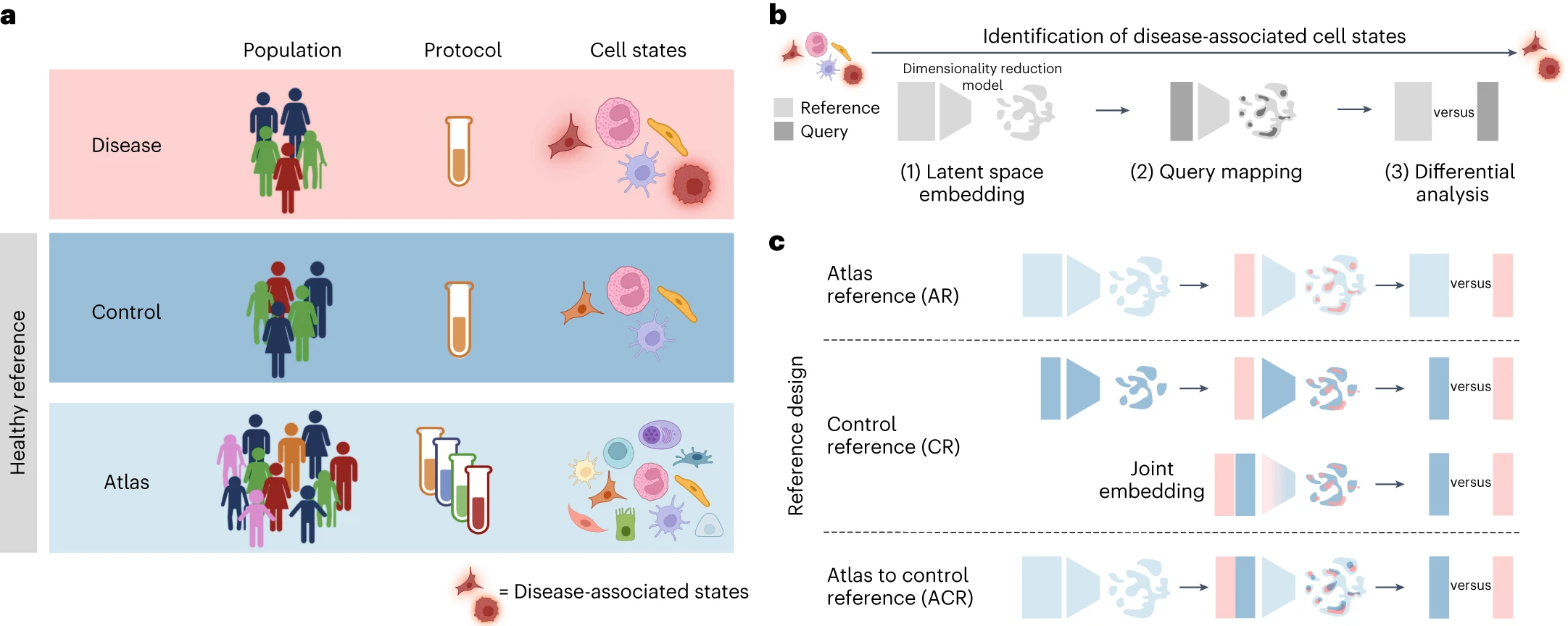
本研究团队评估了单细胞参考图谱的选择如何影响从疾病样本scRNA-seq数据中识别细胞状态变化。利用公开的单细胞转录数据,建立了来自健康个体的单细胞参考图谱,证实使用该图谱进行潜在空间(latent space learning)学习,并对匹配对照进行差异分析,能更好地识别疾病相关细胞,特别是多种受干扰的细胞类型。该研究为设计疾病队列研究和优化细胞图谱的使用提供了指导。
- 论文链接:https://doi.org/10.1038/s41588-023-01523-7
- 代码链接:https://github.com/MarioniLab/oor_benchmark
- 代码链接:https://github.com/MarioniLab/oor_design_reproducibility
博文资讯¶
fanyi包,一个R翻译包,它支持多种翻译源,包括百度,bing,有道等。它可以帮助你在R中进行文本翻译,包括翻译图注,字符串。fanyi包的主要功能是提供了一个translate()函数,它可以接受一个字符串或者一个字符向量作为输入,然后返回一个翻译后的字符串或者字符向量。
- 工具链接:https://github.com/YuLab-SMU/fanyi
 兰雨、汤富酬、周鹏辉、宋春青等人论文获奖。
兰雨、汤富酬、周鹏辉、宋春青等人论文获奖。

本推文通过同源性(homology)、相似性(similarity)、一致性(identity)这个三个易混淆的名字解释了”25%的同源性“的不科学性。
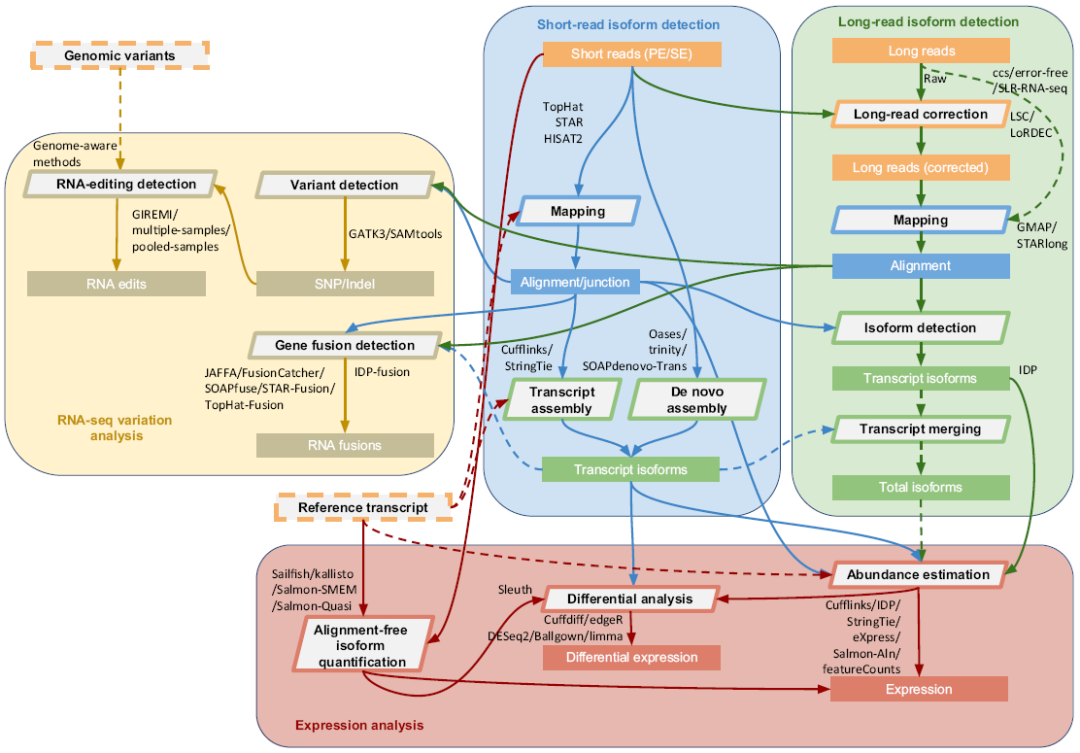
本文详细介绍如何使用 RNA-Seq 技术进行转录组数据的上游分析: RNA-Seq 的原理、流程和常用的分析方法。
工具¶
SCpubr是一个用于单细胞结果可视化的R包,通过它可以绘制出版物级质量的图表。
- 工具链接:https://github.com/enblacar/SCpubr
- 工具教程:https://enblacar.github.io/SCpubr-book/
10、scGPT | 一种新的AI工具预测单细胞中的基因表达

多伦多大学的计算机科学家和细胞生物学家团队BoWang Lab建立了一种新的人工智能(AI)模型,称为单细胞生成式预训练转换器(Single cell generative pretrained transformer, scGPT),可以通过微调来使用scRNA-seq数据执行各种任务。这些任务包括预测操纵特定基因的影响,并将不同批次的数据合并在一起,以揭示否则无法检测到的细胞类型。
- 工具链接:https://github.com/bowang-lab/scGPT
资源¶

本文通过一系列机器学习信息图示,很好地将机器学习的发展历史、关键方法以及未来会如何影响社会生活展现了出来。

来自南方医科大学生物医学大数据研究中心(Bio Med Big Data Center, BMDC)的资源,该中心是专注于生物医学大数据的信息采集、研究分析、提取整合、应用转化的公共服务平台。
历史上的本周¶
- 第68期:颠覆性大滑坡,科研还能有实质创新吗?
贡献者(GitHub ID)¶
「Openbiox 生信周刊」运维小队:
@ShixiangWang(王诗翔)@kkjtmac(阚科佳)@NiEntropy(赵启祥)@He-Kai-fly(何凯)@JnanZhang(张佳楠)@Tomcxf(陈啸枫)@wangdepin(王德品)@kongjianyang(空间阳)
订阅¶
这个周刊每周日发布,同步更新在微信公众号「优雅R」(elegant-r)上。
微信搜索“优雅R”或者扫描二维码,即可订阅。

(完)