生信爱好者周刊(第 39 期):人生不短¶
这里记录每周值得分享的生信相关内容,周日发布。
本杂志开源(GitHub: openbiox/weekly),欢迎提交 issue,投稿或推荐生信相关内容。
封面图¶

本周话题:[人生不短]¶
本周话题来自@ruanyf:人生不短。 人生很短,但是如果你知道,怎么利用好这些时间,它就不短。 人生刚好够实现自己的一个梦想,前提是你必须从一开始,就把所有精力专注于此。 如果你浪费时间,不够专注,那么你没做什么事,人生就结束了。 真正的问题不是人生短暂,而是我们浪费了太多时间。 最令人惊讶的是,人们并不重视自己的时间。你不会让别人偷走你的财产,但你却让别人偷走你的时间。 如果你听任自己为那些不重要的、随机出现的事情分心,那么即使你的寿命有一千年,你也会一事无成。
@NiEntropy -人生是否短暂,每个人都或许会有不同的答案。可或许正如文中所说”人生很短,但是如果你知道,怎么利用好这些时间,它就不短。”人生短暂与否正取决于我们这看不到的财富——时间。
时间就像国王的新衣里面的那件衣服,”聪明人看得到它的宝贵“,于是珍惜起自己所拥有的每分每秒,创造出自己想要的人生。”傻瓜看不到它的价值“挥霍着时间庸庸碌碌度过了他们的一生。
正如现代计算机的发明人冯·诺依曼的故事一样,利用好时间并不只是能让人生变得充实而漫长,更重要的一点是只有这样才能真正做好自己想做的,得到自己想要的。冯·诺依曼的人生短吗?从寿命来说或许不长,但谁能说他的人生不充实呢?他人生的每时每刻都有数不胜数的人需要他的帮助。可对他自己来说呢?直到生命的最后一刻,他是否会后悔,后悔自己最后的时间已经来不及做完自己想做的事呢。后悔于没有把时间用在自己真正想做的事情上呢?
人生短吗?惜时者长,殄时者短!
生信研究¶
1、Genome Biology | 经济实惠的转录组测Prime-seq
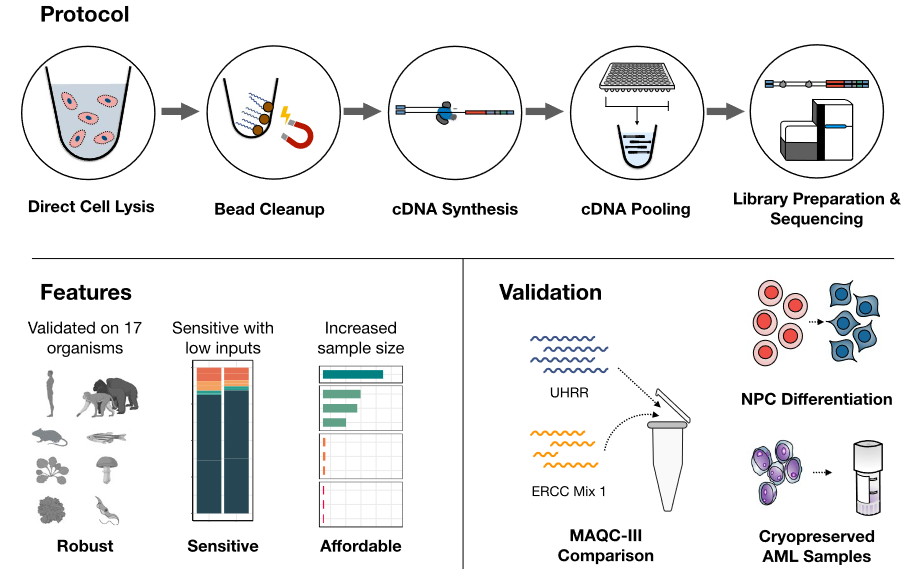 德国慕尼黑大学研究团队研发了一种新的bulk RNA-seq方法——Prime-seq。经验证,Prime-seq与常规的TruSeq的测序数据质量和检测基因数量相似,并且成本更低。
德国慕尼黑大学研究团队研发了一种新的bulk RNA-seq方法——Prime-seq。经验证,Prime-seq与常规的TruSeq的测序数据质量和检测基因数量相似,并且成本更低。
- 论文链接:https://genomebiology.biomedcentral.com/articles/10.1186/s13059-022-02660-8
2、Nature Chemical Biology | 56万条敏感数据系统识别生物标志物驱动的药物组合以克服抗性

理解和预测对治疗药物的应答能力可能会改善癌症患者的预后。作者假设癌细胞系的基础基因转录状态,再加上小分子的细胞活性谱,可用于确定内在抗性的特定机制并预测克服抗性的药物组合,作者分析了 564,424 个敏感性数据谱来识别候选基因-化合物对,并验证了九个这样的关系。
作者确定了一种新关系的机制,其中丝氨酸水解酶单酰基甘油脂肪酶(MGLL)或羧酸酯酶 1(CES1)的表达通过直接酶促修饰赋予了对组蛋白赖氨酸去甲基化酶抑制剂GSK-J4的抗性,不敏感的细胞系可以通过抑制或基因敲除对GSK-J4敏感。
-
论文链接:https://www.nature.com/articles/s41589-022-00996-7
-
代码地址: https://github.com/broadinstitute/prism_data_processing https://github.com/reesmg
3、Nature Genetics | 基于单细胞转录组数据绘制小鼠胚胎发育轨迹新图谱,全面解析胚胎发育细胞状态
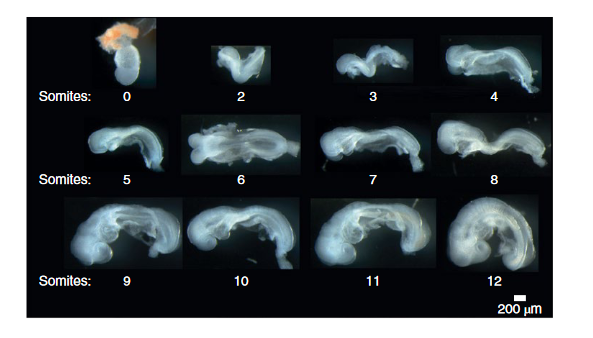 美国华盛顿大学医学院的研究团队,利用单细胞转录组测序技术系统地重构了小鼠胚胎发育的细胞轨迹图,为理解完整的胚层谱系建立及细胞的命运调控机制提供了数据与思路。
美国华盛顿大学医学院的研究团队,利用单细胞转录组测序技术系统地重构了小鼠胚胎发育的细胞轨迹图,为理解完整的胚层谱系建立及细胞的命运调控机制提供了数据与思路。
- 文章链接:https://www.nature.com/articles/s41588-022-01018-x
4、Nature Reviews Cancer | 肿瘤蛋白质基因组学研究进展及未来前景
 Broad研究所发表了关于蛋白质基因组学在肿瘤研究中的最新进展和关键发现的综述性文章,文章描述了蛋白质基因组学中样本收集技术以及生成、分析和整合蛋白质组学与基因组数据的方法等。进行此类研究主要基于在肿瘤研究中,以现有的研究水平鉴别驱动突变尚存问题,诸如单个肿瘤中复发性点突变、小片段插入或缺失的数量较少,色体拷贝数扩增或缺失,靶向治疗耐药性的频繁发展等等我们难以预知后果或是难以建模的问题对于肿瘤的研究带来了极大的困难。
为了更好的克服这些困难,一些免疫肿瘤学方法迅速发展,其中蛋白质组学更是可以在肿瘤微环境与肿瘤细胞的信号转导层面提供更为具象的信息。蛋白质组学技术的目的是识别肿瘤中变化的蛋白质和PTM,并对该数据进行量化,随后与拷贝数变异、表观遗传沉默、mRNA表达的变化联系起来以提供有关蛋白质调节和响应这些变化的信号的直接信息。基于MS的蛋白质组学还提供了对PTM的深入定量分析。
因此,蛋白质基因组学有可能为疾病治疗提供重要见解,并在临床环境中用于诊断多种耐药信号及其PTM,并将研究重点集中在可立即用于药物开发的靶点上,或集中精力开发新疗法。
Broad研究所发表了关于蛋白质基因组学在肿瘤研究中的最新进展和关键发现的综述性文章,文章描述了蛋白质基因组学中样本收集技术以及生成、分析和整合蛋白质组学与基因组数据的方法等。进行此类研究主要基于在肿瘤研究中,以现有的研究水平鉴别驱动突变尚存问题,诸如单个肿瘤中复发性点突变、小片段插入或缺失的数量较少,色体拷贝数扩增或缺失,靶向治疗耐药性的频繁发展等等我们难以预知后果或是难以建模的问题对于肿瘤的研究带来了极大的困难。
为了更好的克服这些困难,一些免疫肿瘤学方法迅速发展,其中蛋白质组学更是可以在肿瘤微环境与肿瘤细胞的信号转导层面提供更为具象的信息。蛋白质组学技术的目的是识别肿瘤中变化的蛋白质和PTM,并对该数据进行量化,随后与拷贝数变异、表观遗传沉默、mRNA表达的变化联系起来以提供有关蛋白质调节和响应这些变化的信号的直接信息。基于MS的蛋白质组学还提供了对PTM的深入定量分析。
因此,蛋白质基因组学有可能为疾病治疗提供重要见解,并在临床环境中用于诊断多种耐药信号及其PTM,并将研究重点集中在可立即用于药物开发的靶点上,或集中精力开发新疗法。
- 论文链接: https://www.nature.com/articles/s41568-022-00446-5
博文资讯¶
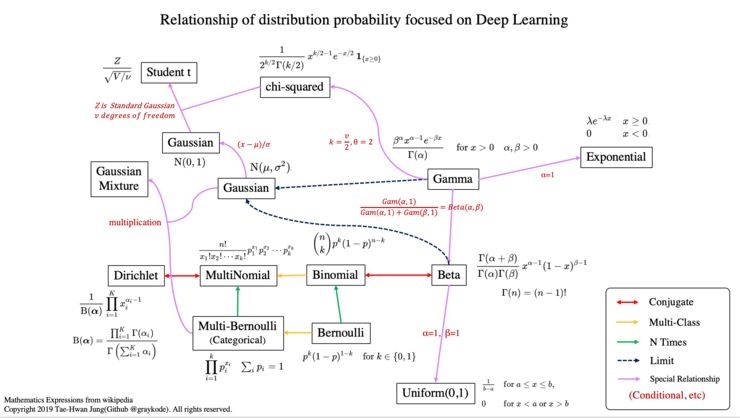
文章首先对概率分布进行概述,随后介绍了与深度学习相关的13种概率分布及其特征,并提供github学习链接。 - 论文链接:https://www.nature.com/articles/nature25501 - R包介绍:http://research-pub.gene.com/IMvigor210CoreBiologies/
 肿瘤细胞的全身扩散是大多数癌症死亡的最终原因,但很少有成功的治疗策略专门针对转移。作者介绍了癌症转移的过程及类型、目前治疗方法的局限性以及未来的治疗前景。
肿瘤细胞的全身扩散是大多数癌症死亡的最终原因,但很少有成功的治疗策略专门针对转移。作者介绍了癌症转移的过程及类型、目前治疗方法的局限性以及未来的治疗前景。
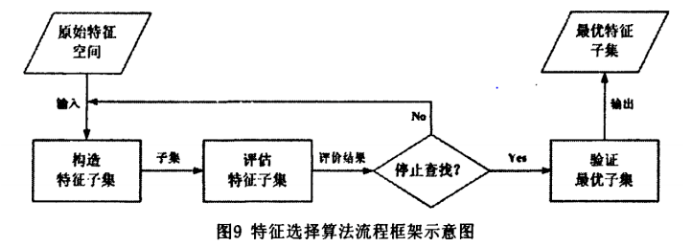
特征选择也被成为变量选择、属性选择或者变量子集选择,是指为了构建模型而选择相关特征(即属性、指标)子集的过程。建模过程中太多的特征会增加模型的复杂性和过拟合,而太少的特征会导致模型的拟合不足,因此将模型优化为足够复杂以使其性能可推广,但又足够简单易于训练、维护和解释是特征选择的主要工作。本文针对一些常见的特征选择策略进行概述。
工具¶
8、InterpretML - 可说明的模型,解释黑箱机器学习
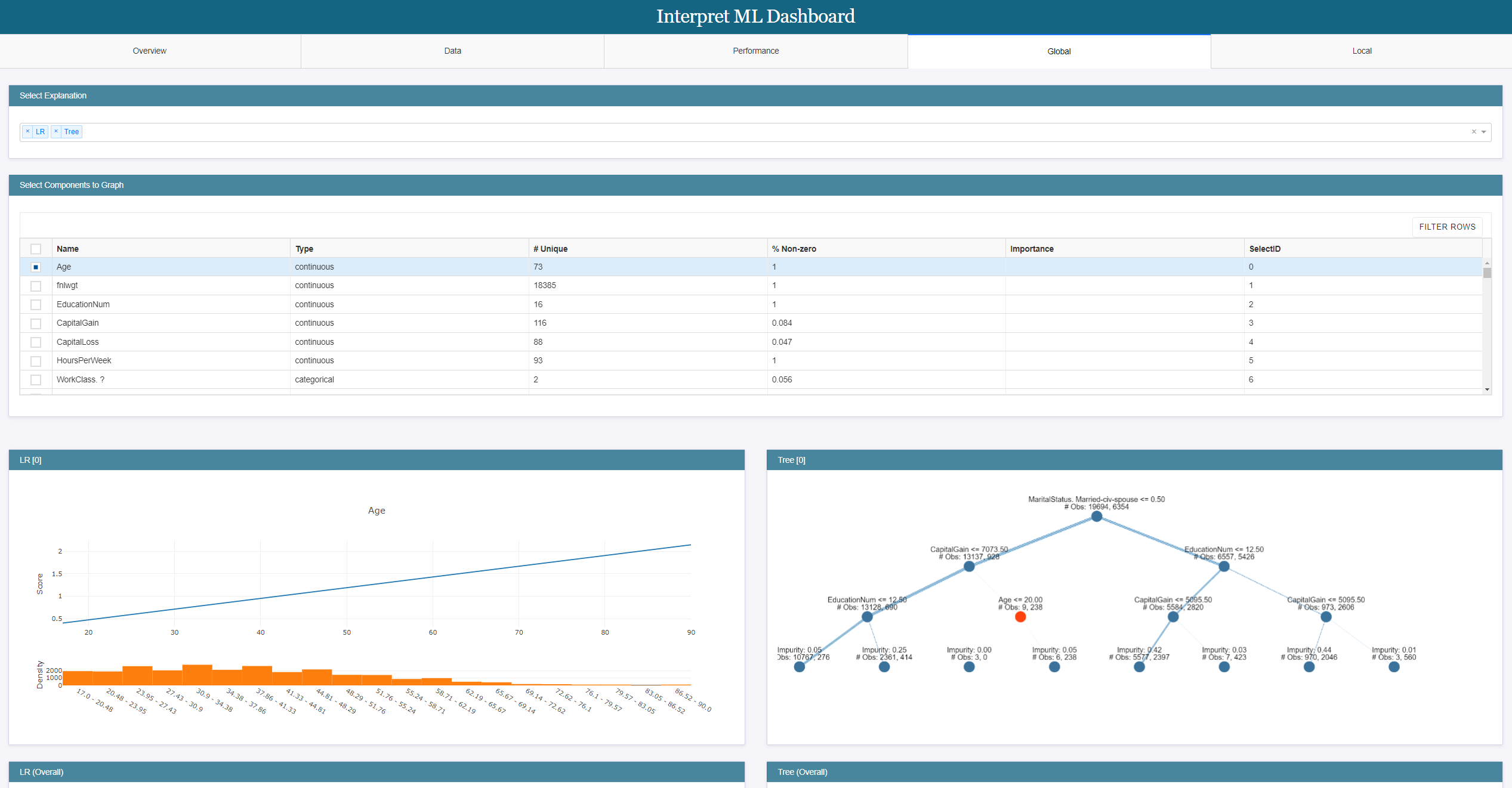
InterpretML是一个开源包,它将最先进的机器学习可解释性技术整合在一个框架下。使用这个包,你可以训练可解释的glassbox模型和解释黑箱系统。InterpretML可以帮助你理解模型的全局行为,或者理解单个预测背后的原因。

-
文档:https://interpret.ml/docs/intro.html
-
示例:https://nbviewer.org/github/interpretml/interpret/blob/master/benchmarks/EBM%20Classification%20Comparison.ipynb

grateful的目标是使在任何分析中引用R和R包变得非常容易,这样包的作者就得到了他们应得的荣誉。通过调用一个函数,grateful将扫描项目中使用的R包,并生成包含这些包的所有引用的BibTeX文件。然后grateful就可以生成一个新的文档,其中包含所需输出格式的引用(Word、PDF、HTML、Markdown)。这些参考文献可以针对特定的期刊进行格式化,这样我们就可以直接将它们粘贴到我们的手稿或报告中。
10、cnv_facets

cnv_facets 包利用全基因组 (WGS)、全外显子组 (WEX) 和靶向(panel)测序得到的NGS数据,检测体细胞拷贝数变异 (CNV)。相较于传统的facets包,其优化了从bam到vcf的步骤。
- 代码地址: https://github.com/dariober/cnv_facets
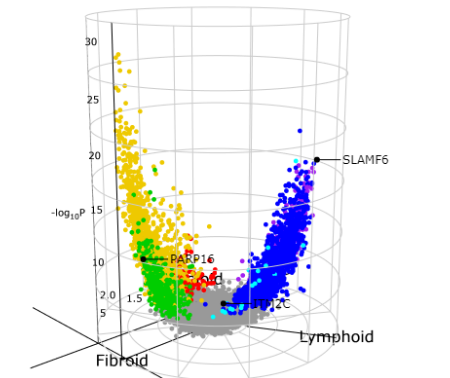
volcano3D包开发的主要目的是在三维火山图上可视化差异表达基因的位置,并可利用plotly转化为交互动态格式的网页文件,展现形式令人耳目一新,同时利用R shiny app将该工具部署在PEAC网址上面。
- 代码地址: https://github.com/KatrionaGoldmann/volcano3D

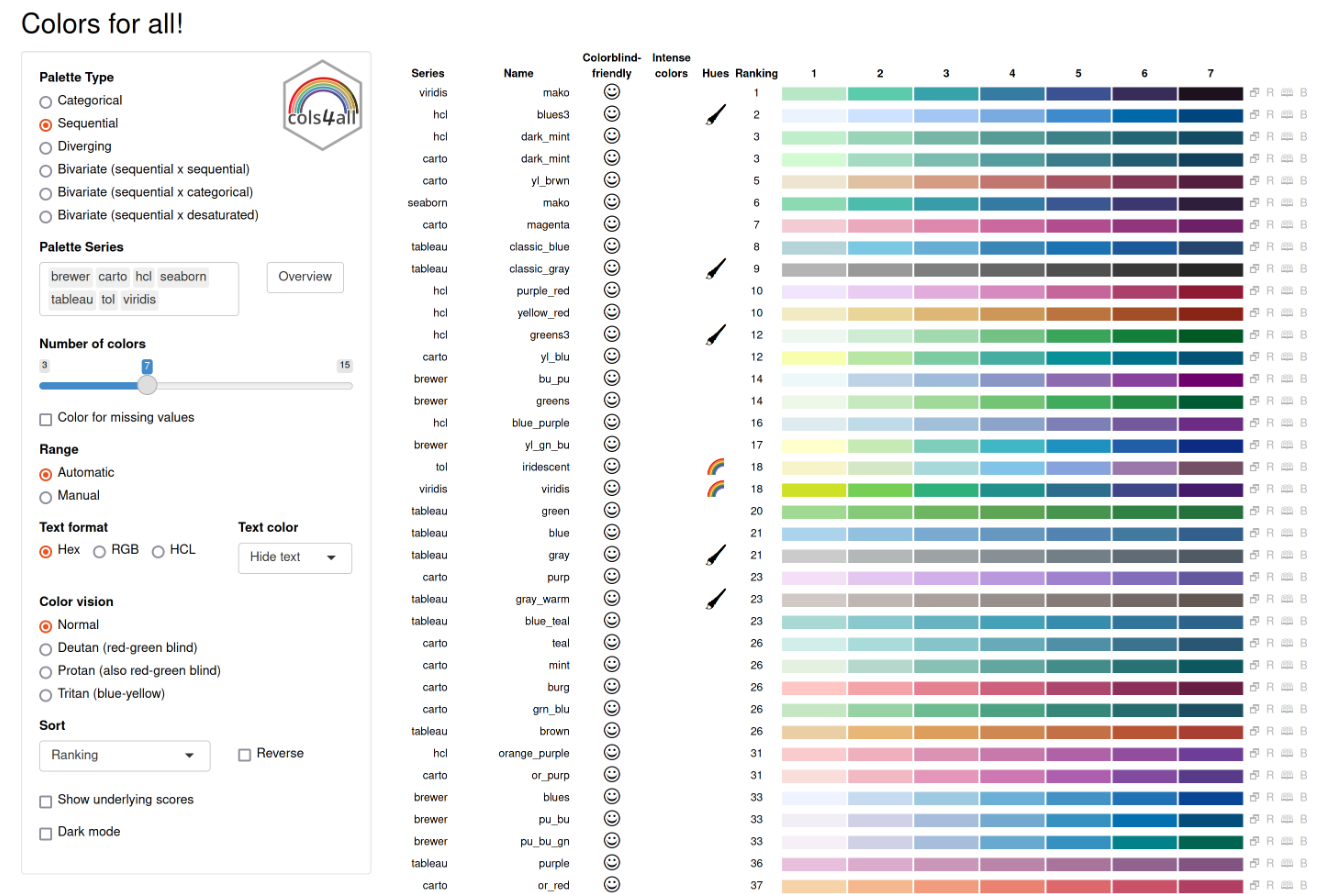
- R包地址: https://github.com/mtennekes/cols4all
资源¶
13、r-manuals
该网址为R manual的重新设计便于搜索的新版本,由R开发核心团队维护与支持,尽量保持与原版本贴近的内容,涵盖了R语言简介、数据导入/导出、安装和管理、R扩展、语言定义及内部结构几方面的内容。
-
手册: https://cran.r-project.org/manuals.html
-
代码地址: https://github.com/rstudio/r-manuals
14、全肿瘤靶向药物全景图
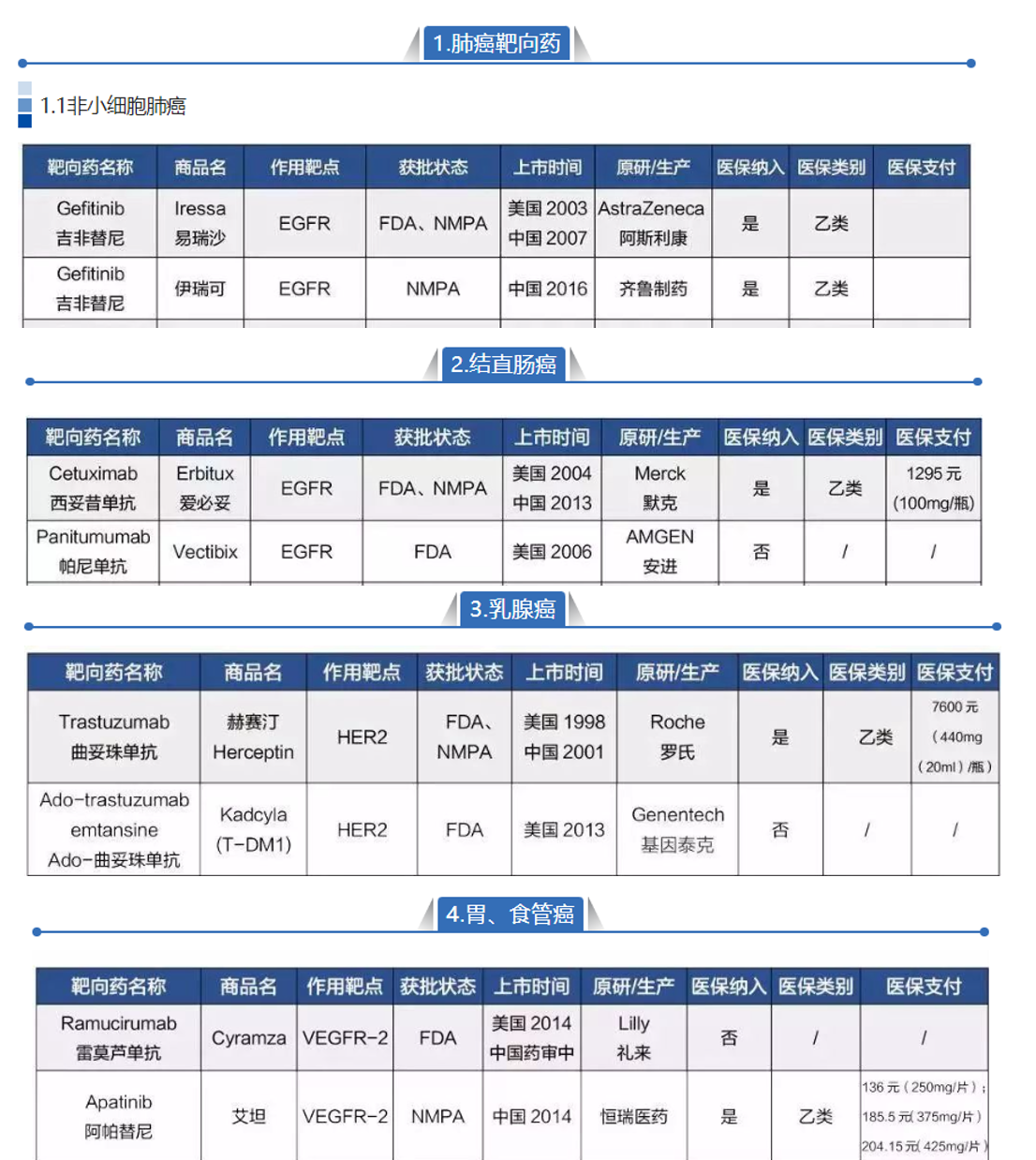 汇总了34种癌症的靶向治疗药物信息。
汇总了34种癌症的靶向治疗药物信息。
- 文章链接: https://mp.weixin.qq.com/s/0tMlcEppkirpcnOTMnEzUA
15、MOFA
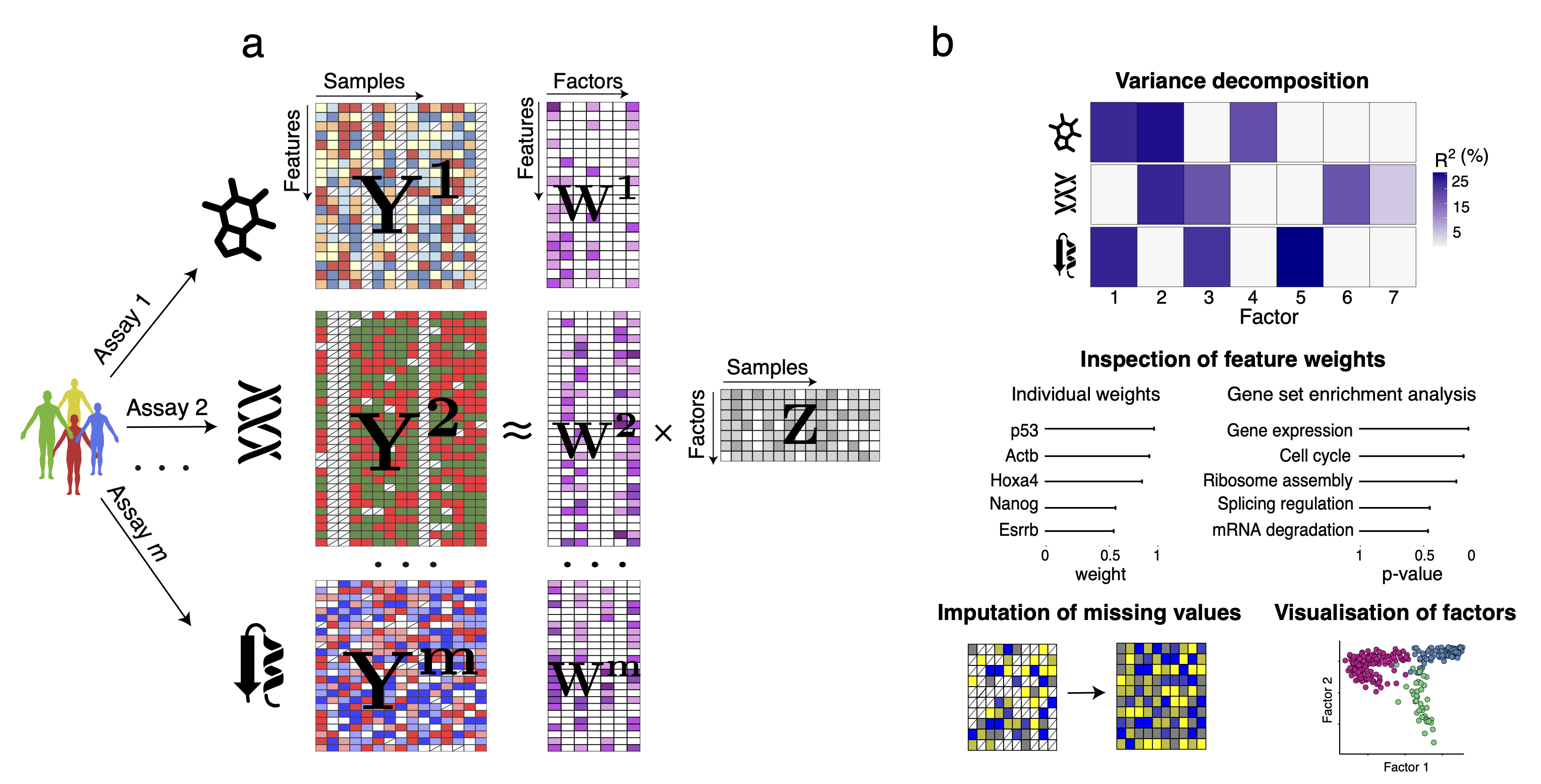
MOFA(多组学因素分析)是一个因子分析模型,它为通过无监督方式集成的多组学数据集提供了一个通用框架。
- 链接: https://biofam.github.io/MOFA2/
贡献者(GitHub ID)¶
「Openbiox 生信周刊」运维小队:
@ShixiangWang@kkjtmac@NiEntropy@He-Kai-fly@JnanZhang@Tomcxf@wangdepin
订阅¶
这个周刊每周日发布,同步更新在微信公众号「优雅R」(elegant-r)上。
微信搜索“优雅R”或者扫描二维码,即可订阅。

(完)