生信爱好者周刊(第 51 期):职业对性格的改变¶
这里记录每周值得分享的生信相关内容,周日发布。
本杂志开源(GitHub: openbiox/weekly),欢迎提交 issue,投稿或推荐生信相关内容。
封面图¶

本周话题:职业对性格的改变¶
本周话题来自@ruanyf:职业对性格的改变,人类行为受到工作环境的强烈影响,你会变成环境要求你成为的样子。很多人都有一种印象,会计师的性格是斤斤计较、对数字格外敏感,律师的性格是重视利益最大化,海关执法人员的性格则是充满怀疑、不信任他人,这就是职业对性格的改变。程序员这个职业,会如何改变一个人的性格?你会变得习惯孤军奋斗,靠自己解决问题。
@NiEntropy - 科研对性格有什么改变?科研是一个探索未知、不断创新的工作,既要脚踏实地,又要天马行空。科研对于我大概就像做梦吧,祝大家美梦成真!
生信研究¶
1、Nature Methods | NEAT-seq在单细胞水平同时检测转录因子、染色质可及性及转录组

由斯坦福大学团队开发的新测序技术NEAT-seq可以对单个细胞同步定量核蛋白、染色质可及性和转录组,进而从表观层面探索转录因子、染色质状态和基因表达直接的关系。
- 论文链接:https://www.nature.com/articles/s41592-022-01461-y
2、Nat Cell Biol | 生物大数据分析揭示全新的基因调控模式和潜在的抗癌药物靶点

研究团队基于对来自10种不同癌症类型,超过8000个转录组测序样本的数据分析,文章揭示了一种以往被忽视的基因调控模式——位于3’端非编码区域的剪接,在癌细胞中普遍存在,并显著上调来驱动肿瘤发生。通过对这些大批量测序数据的分析,作者们在不同癌症病人样本中鉴定出来大量的3’端非编码区域剪接事件,其中包括超过1000个常见的事件可以在超过半数的病人样本中发现。尤其是在肝细胞癌(LIHC)以及结肠癌(COAD)病人中,更多的3’端非编码区域的剪接和病人更短的生存时间显著相关。作者们将这超过1000种不同的剪接事件在10种癌症的丰度,以及它们和病人总体生存时间的相关性都收集到一个数据库——SpUR,以供研究人员访问和使用。
- 论文链接:https://www.nature.com/articles/s41556-022-00913-z
3、Understudied Proteins Initiative | 未充分研究蛋白质计划
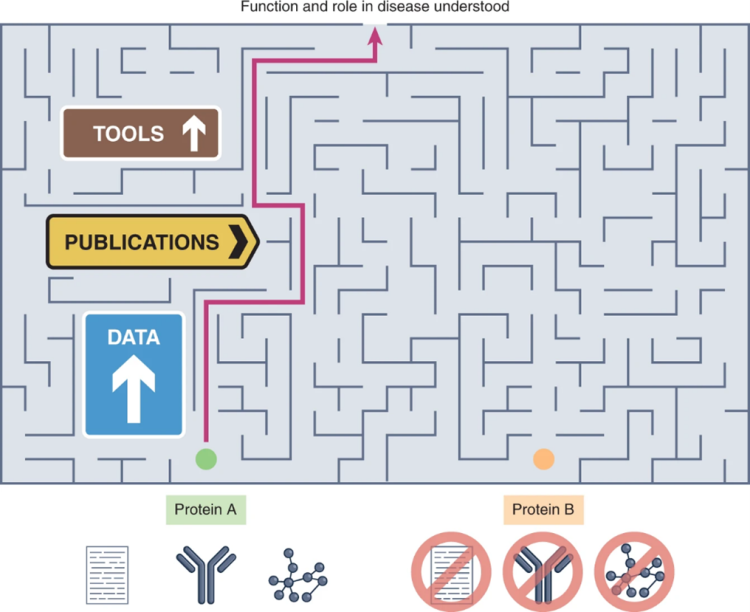
迄今为止,大多数研究都集中在有限的一组越来越知名的蛋白质上,而数千种人类蛋白质的生物学功能仍然知之甚少。这种注释偏差也被称为 “路灯效应” 或 “富人-富人综合征”,严重阻碍了生物医学的发展。基因疾病之间的关联机制研究通常集中在已经众所周知的蛋白质上,与此同时,许多与疾病相关的未表征的蛋白质没有经过功能研究。
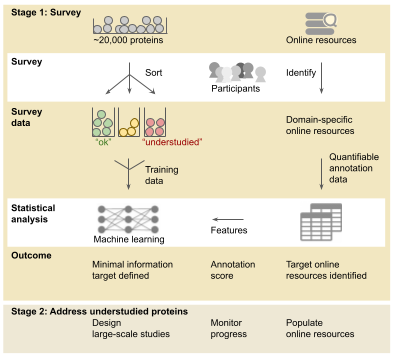
六国科学家联合发起“未充分研究蛋白质计划”,这项调查的目的是让我们更好地了解哪些人类蛋白质仍然被低估,以及为什么。
- 调查问卷:https://understudiedproteins.org/survey
- 论文链接:
https://pubmed.ncbi.nlm.nih.gov/35534633/ https://pubmed.ncbi.nlm.nih.gov/35534555/
4、Nature Medicine | 基于群体学习的分散式人工智能在癌症组织病理学中的应用

本研究展示了群体学习(SL)在5000多名患者的千兆像素组织病理学图像的大型多中心数据集中上的成功应用。作者表明,使用SL训练的人工智能(AI)模型可以直接从结直肠癌H&E染色的病理切片上预测BRAF突变状态和微卫星不稳定性。作者在北爱尔兰、德国和美国三类患者人群中训练AI模型,并在来自英国的两个独立数据集中验证了预测性能。数据显示,经过SL训练的AI模型优于大多数本地训练的模型,并与在合并数据集上训练的模型表现相同。此外,作者展示了基于SL的AI模型是数据高效的。未来,SL可用于训练分布式AI模型,用于任何组织病理学图像分析任务,从而无需数据传输。
- 论文链接:https://doi.org/10.1038/s41591-022-01768-5
博文资讯¶

ROC曲线和曲线下面积AUC被广泛用于评估二元分类器的性能。但是有时,基于精确召回曲线下面积 (AUPRC) 的测量来评估不平衡数据的分类却更为合适。本文详细比较这两种测量方法,并说明在AUPRC数据不平衡的情况下衡量性能时的优势。
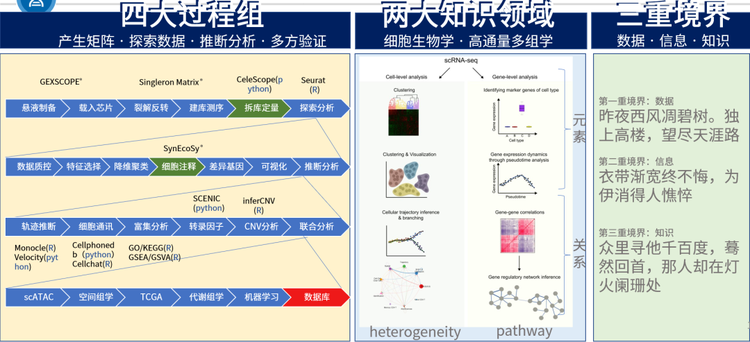
单细胞大佬对单细胞数据科学研究的感悟:四大过程——产生矩阵,探索数据,推断分析,多方验证; 两大领域——细胞生物学、高通量多组学;三重境界——数据、信息、知识。
该文展示了一个通用性的GEO芯片处理流程,能够解决80%以上的芯片数据处理问题。包括数据的下载,ID转换和清洗,质控以及差异分析等。
8、clusterProfiler | KEGG的富集分析终于真的本地化了
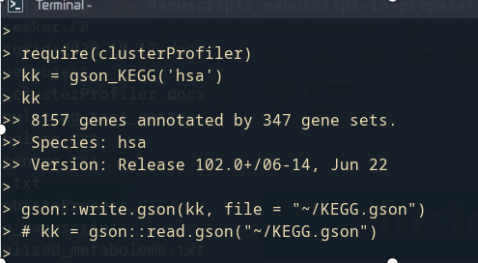
本文介绍了使用clusterProfiler进行本地化 KEGG富集分析的方法(终于不用联网等待了~)。
工具¶
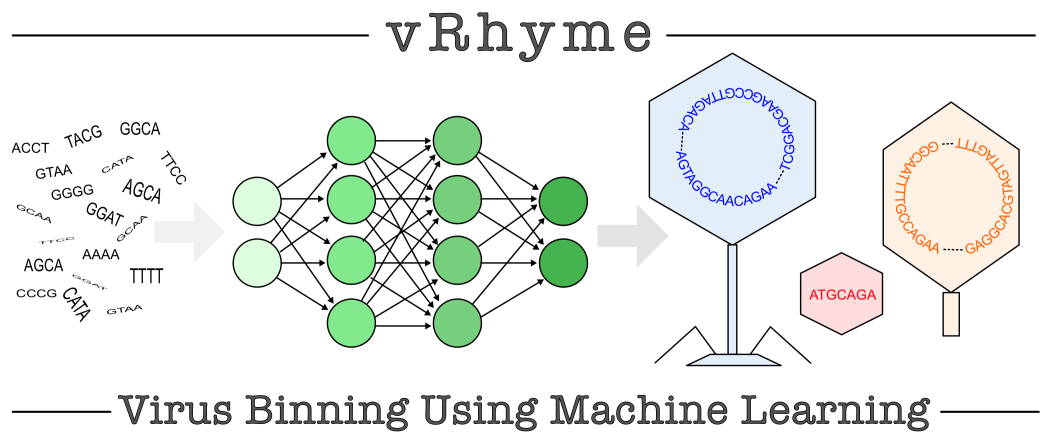
基因分箱对于研究细菌和古菌非常重要,有大量的细菌和古菌的分箱工具,vRhyme首次提供了从宏基因组中分离病毒基因组的方案。vRhyme的工作流程分为五个步骤:read覆盖率处理、序列特征提取、有监督的机器学习、迭代网络聚类和bin评分。
- 论文链接:https://academic.oup.com/nar/article/50/14/e83/6584432
- Github:https://github.com/AnantharamanLab/vRhyme
10、SCDC | 基于多个单细胞RNA测序参考的bulk基因表达反卷积
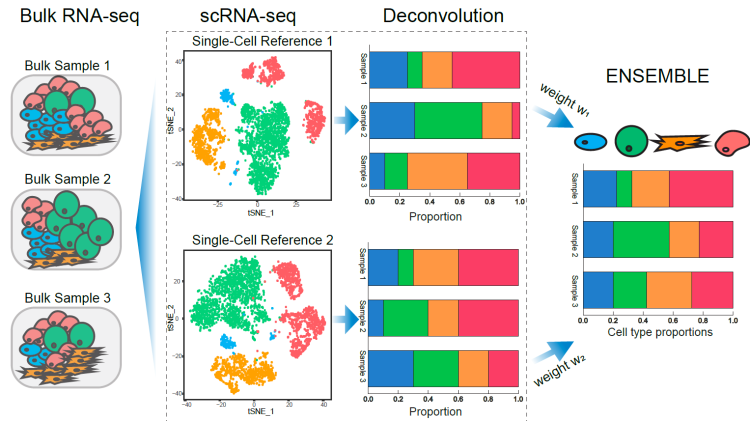
SCDC是一种用于bulk RNA-seq的反褶积方法,它利用来自多个单细胞参考数据集的细胞类型特定基因表达。SCDC采用ENSEMBLE方法集成来自不同实验室和不同时间产生的不同scRNA-seq数据集的反褶积结果,隐式解决了批处理效应的混杂。
- 论文链接:https://doi.org/10.1093/bib/bbz166
11、scDIOR | 打通R和python两大平台的单细胞数据转换工具
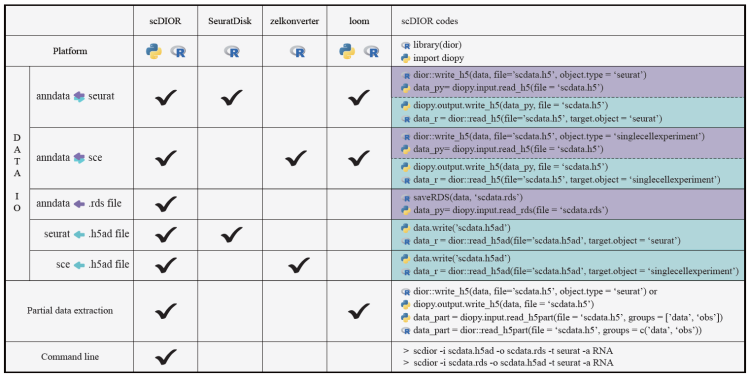
scDIOR是一个基于HDF5将单细胞数据在R和Pyhton两大平台下进行数据转换的工具。
- 论文链接:https://doi.org/10.1186/s12859-021-04528-3
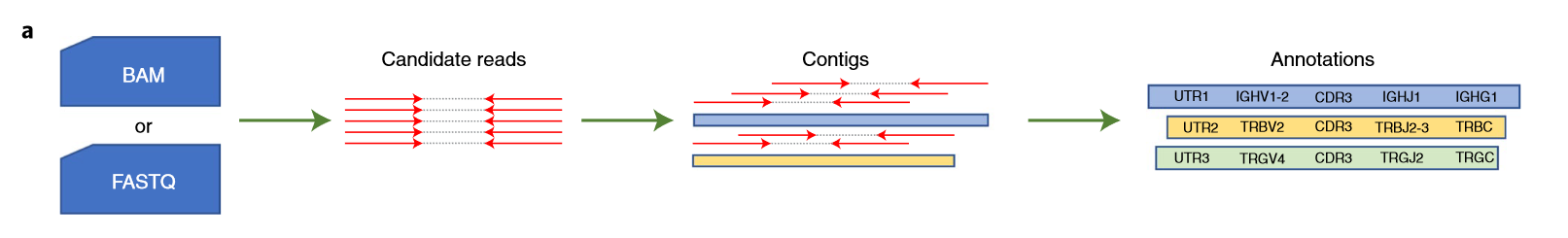
由刘小乐课题组中Li Song等人开发的TRUST4算法,一种用于bulk RNA-seq和单细胞RNA-seq数据中识别免疫细胞受体序列的算法。TRUST4对受体基因进行从头组装,构成重叠群。然后将组装好的重叠群与IMGT参考基因序列重新比对,鉴定出重叠群中的V,D,J以及C基因。TRUST4支持具有任何读段长度的单端和双端测序数据。
- Github:https://github.com/liulab-dfci/TRUST4
- 论文链接:https://www.nature.com/articles/s41592-021-01142-2
资源¶
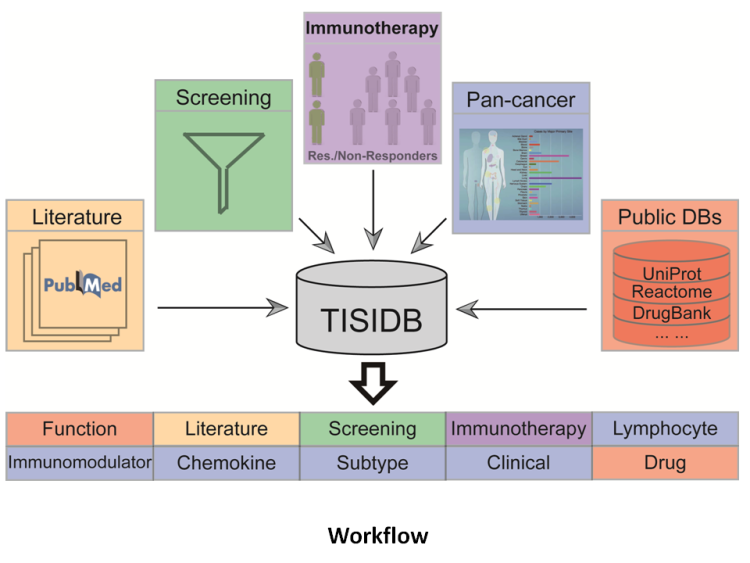
TISIDB是一个肿瘤和免疫系统相互作用的数据库,它集成了多种异构数据类型:PubMed文献挖掘、TCGA、UniProt、GO、DrugBank等。
- 链接:http://cis.hku.hk/TISIDB/index.php
- 论文链接:https://academic.oup.com/bioinformatics/article/35/20/4200/5418799?logn=false

HeatmapR包中通过mtcars数据展示了如何使用本工具快速绘制复杂热图。
- 教程:https://dillonhammill.github.io/HeatmapR/
汇总了scRNA-seq数据分析(Seurat包学习笔记、Scater包学习笔记、Monocle2包学习笔记、Palantir包学习笔记)、scATAC-seq数据分析(Signac包学习笔记、Cicero包学习笔记)和单细胞类型分类注释分析。
历史上的本周¶
贡献者(GitHub ID)¶
「Openbiox 生信周刊」运维小队:
@ShixiangWang(王诗翔)@kkjtmac(阚科佳)@NiEntropy(赵启祥)@He-Kai-fly(何凯)@JnanZhang(张佳楠)@Tomcxf(陈啸枫)@wangdepin(王德品)
订阅¶
这个周刊每周日发布,同步更新在微信公众号「优雅R」(elegant-r)上。
微信搜索“优雅R”或者扫描二维码,即可订阅。
 (完)
(完)