生信爱好者周刊(第 48 期):人生不能只有一个支点¶
这里记录每周值得分享的生信相关内容,周日发布。
本杂志开源(GitHub: openbiox/weekly),欢迎提交 issue,投稿或推荐生信相关内容。
封面图¶

超过四成的电脑仍然无法升级到 Windows 11。via
本周话题:人生不能只有一个支点¶

科研是一份没有上下班,几乎全天候头脑在线的工作,对于需要整天泡实验室的科研人而言,想工作之余管管老人孩子更是分身乏术,因此诸多优秀的(女)博士不得不在人生的岔路口选择退出学术道路。
@ShixiangWang - 对于科研职业道路来说,当前的环境下博士只是起点。随着我国社会进程在科技、经济、教育等多方面的发展,越来越多的学生涌入研究生教育与学术道路,不平等、压力、不正常的师生关系、不正常的人才职业化培养政策等等都会在这个环境下进行交互,塑造每个人在不同情景下的独特经历。就像高中老师在告诉我们“大学就可以自由、放飞自我”一样,我们应该意识到自我应该有着更多的价值判断并付诸行动,“你的人生只能是你的人生,别人并不会过多的关注”,人生不能只有一个支点,就像你单腿无法正常行走。科研工作只能是人生的一部分(也许的确有人乐意去把科研与人生等同对待,但也需要意识到科研归属于人生,而不是相反的逻辑,即人生永不可能归属于科研),那些灌输人生只有科研或者类似奉献的言语与文化只是一种欺骗,灌输者从不会奉行。切记每个人都独一无二,存在着比你想象更多的可能性。希望人间少一些白发人送黑发人,至少做科研的人生最后不应该停留在这里。
生信研究¶
1、Nature | 人类泛基因组计划:更完整、更准确的人类参考基因组
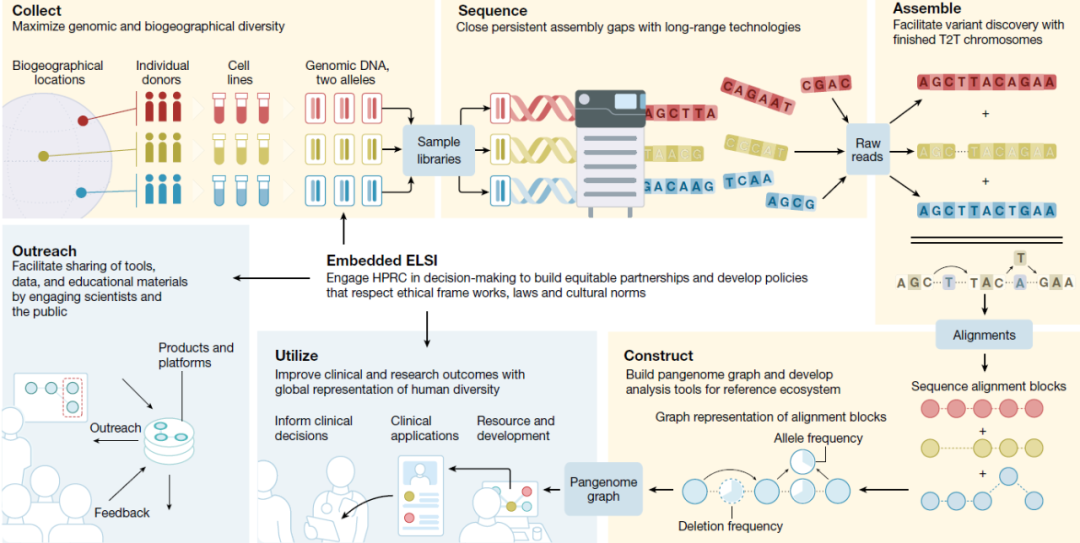
人类泛基因组参考联盟(Human Pangenome Reference Consortium, HPRC) 旨在创建一个更为精准和完整的人类参考基因组,该计划以图形为基础、以端粒到端粒的测序结果建立尽可能高质量的人类参考泛基因组。近日,HPRC在Nature上发表了题为“The Human Pangenome Project: a global resource to map genomic diversity”的观点文章,概述了人类泛基因组参考联盟的战略目标、挑战和机遇。希望与全球范围内的科学家和生物伦理学家共同创建一个完整的人类全基因组参考资源,使其更多地涵盖整个基因组范围内的变异信息,为更大规模人类基因组资源提供终极指南,为开发新的概念框架和分析方法,以构建下游分析和可视化的全基因组基础设施和工具奠定坚实基础。
- 论文链接:https://www.nature.com/articles/s41586-022-04601-8
2、Nature | 基因变异、表达与疾病:单细胞表达数量性状基因座(eQTL)
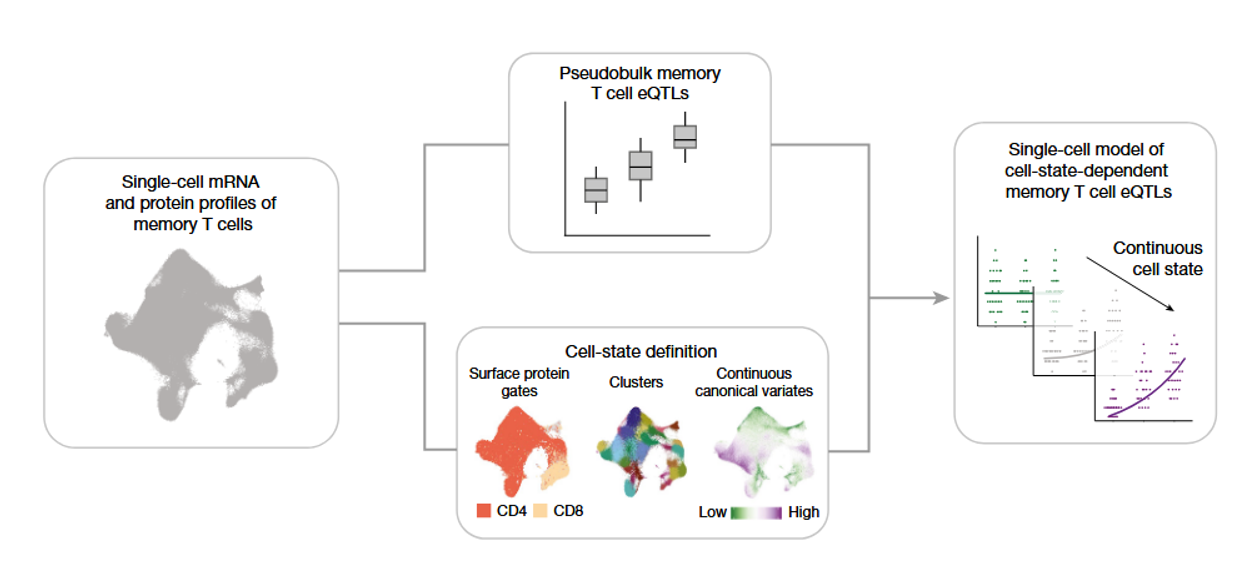
所有能够度量的性状都可以称为数量性状(QT),这些性状呈连续的,不可以严格的分类,而数量性状基因座(QTL)即控制数量性状的基因在基因组中的位置,而表达数量性状基因座(eQTL)就是能控制数量性状基因的表达水平高低的基因在基因组中的位置。近日,美国布莱根妇女医院与哈佛医学院Soumya Raychaudhuri研究组在Nature上发表了文章,聚焦记忆T细胞,在单细胞分辨率水平建立了的eQTL模型,捕获细胞状态依赖的基因调控变异。
- GitHub:https://github.com/immunogenomics/sceQTL
- 论文链接:https://www.nature.com/articles/s41586-022-04713-1
3、Nature Methods | 系统评估16种空间转录组和单细胞转录组数据整合算法在预测基因或细胞类型空间分布方面的性能

目前空间和单细胞转录组数据整合算法已经非常丰富了,但是还未有系统的研究来评估它们在分析时的具体表现。因此中国科学技术大学生命科学与医学部瞿昆教授课题组在Nature Methods上发表的这篇文章,通过45对同一组织来源的空间转录组与单细胞转录组数据集,结合32个模拟数据集,并设计了多种指标,从准确性、鲁棒性、计算资源耗时等多维度系统性评估了16种整合算法在预测基因或细胞类型空间分布方面的性能。
- 论文链接:https://www.nature.com/articles/s41592-022-01480-9
- 论文代码:https://github.com/QuKunLab/SpatialBenchmarking
4、Science Advances | NK细胞可通过胞啃作用获取肿瘤细胞的PD-1,失去抗癌能力

本研究发现NK细胞表面存在的免疫检查点PD-1并非来自内源性表达,而是NK细胞通过由SLAM受体介导的胞啃作用,从肿瘤细胞的细胞膜上获取的。得到PD-1后,NK细胞的抗肿瘤免疫功能被抑制,不过可以由PD-1抑制剂挽救。
- 论文链接:https://www.ncbi.nlm.nih.gov/pmc/articles/PMC9007500/
博文资讯¶
为了全局性的设置ComplexHeatmap的字体参数,我们可以先创建一个全局的viewport,在其中设置某种字体,然后让ComplexHeatmap的热图位于其中。这样一来,所有heatmap中的字体参数都将继承这个全局viewport中设置的字体参数。
- ComplexHeatmap包:https://bioconductor.org/packages/release/bioc/html/ComplexHeatmap.html

- 教程:https://cran.r-project.org/web/packages/regplot/regplot.pdf

来自科技爱好者周刊(第202期)的周话题-三个有启发的学习方法:
- 对重要内容记笔记,并尽量记住。
- 对旧事物进行大量练习,在前人研究的基础上做出新的发现。
- 努力学习你最感兴趣的东西。
8、finalfit包,简单快速输出单因素和多因素结果,并可视化模型结果

finalfit 包,在建模时快速创建精美的结果表和绘图,支持多种表格:cross table、modle table、survey table等,可以轻松导出为 Word 文档、PDF 或 html 文件。
- 教程:https://finalfit.org/articles/finalfit.html
工具¶
dm填补了单个数据框和关系数据库之间数据管道的空白。它是一种联接表语法,为使用、创建和部署关系数据模型提供了一组一致的动词。对于单个研究人员来说,它拓宽了他们可以使用的数据集的范围以及如何使用这些数据集。对于组织来说,它使团队能够快速高效地创建和共享大型、复杂的数据集。

pup是用于处理HTML的命令行工具。它从stdin读取,打印到stdout,并允许用户使用CSS选择器过滤页面的部分。受jq这种JSON处理器的启发,pup的目标是成为探索终端HTML的快速灵活的方式。
11、methylKit - 甲基化注释和分析的R语言工具包
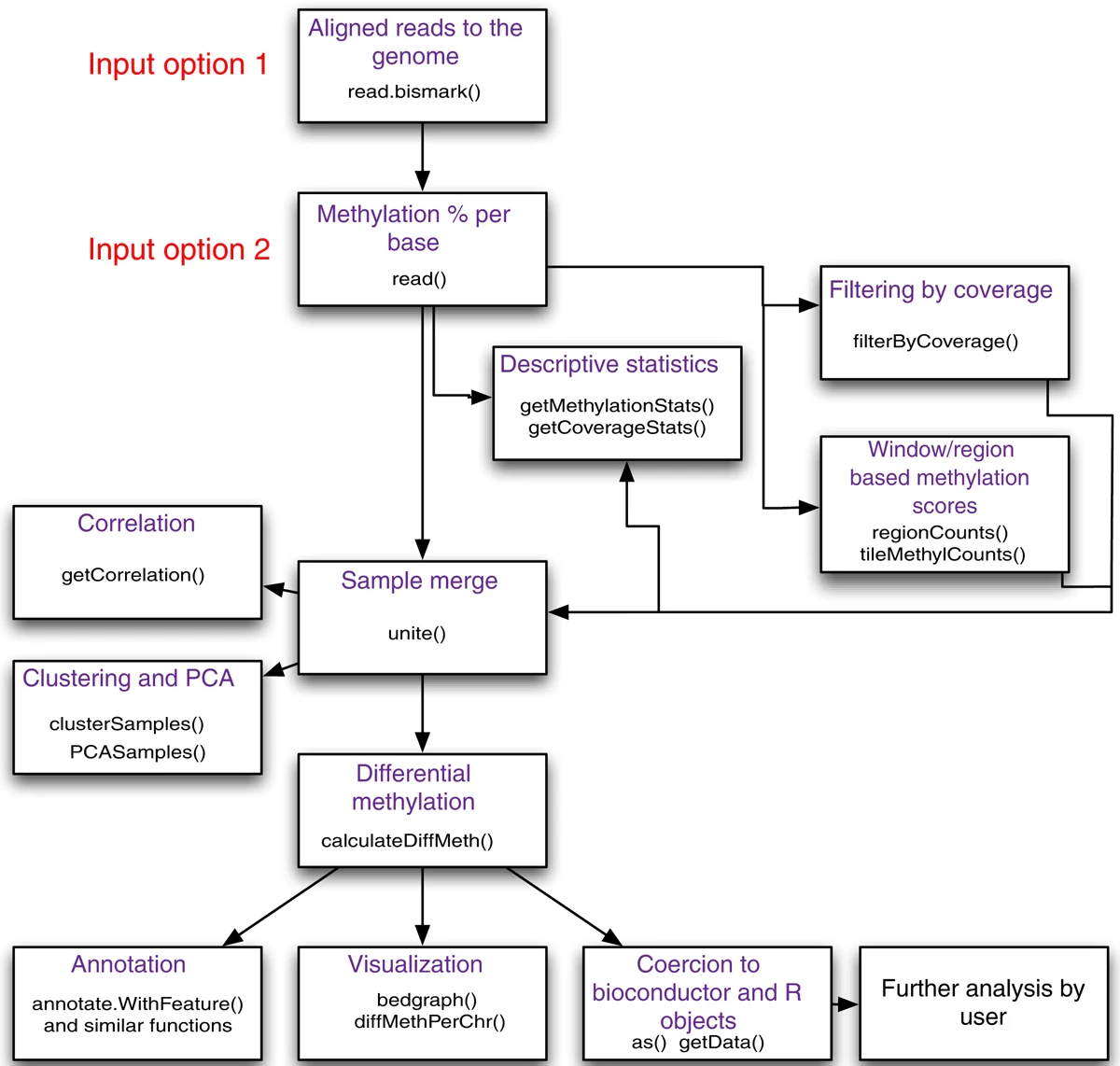
methylKit是一个对甲基化测序数据进行质控、聚类、差异分析和注释等多种功能分析的R包。
- 论文链接:https://genomebiology.biomedcentral.com/articles/10.1186/gb-2012-13-10-r87
12、futile.logger - 类似 log4j 的R日志包
flog.info("Hello, %s", "world")
# Put pid in logging in multi-processing
flog.info('%d message', Sys.getpid())
# This won't print by default
flog.debug("Goodbye, %s", "world")
# Change the log level to debug and try again
flog.threshold(DEBUG)
flog.debug("Goodbye, %s", "world")
# Keep an alternate logger at WARN
flog.threshold(WARN, name='quiet')
# This won't print since it's using the logger named 'quiet'!
flog.debug("Goodbye, %s", "world", name='quiet')
SARTools是一个R包,专门用于RNA-seq数据的差异分析。它提供了一些工具来生成描述性和诊断图,使用著名的DESeq2或edgeR包运行差异分析,并将结果导出到易于阅读的以制表符分隔的文件中。它还有助于生成HTML报告,该报告显示所生成的所有数字,解释统计方法并给出差异分析的结果。注意SARTools并不打算取代DESeq2或edgeR:它只是提供了一个与之配套的环境。关于DESeq2或edgeR背后方法的更多细节,用户应该阅读它们的文档和论文。
在线文档:https://colab.research.google.com/drive/1hoPcImQkct0yPz5nnYcJFOOX9O0EHjtB?usp=sharing#scrollTo=j3IvZ98bji7x
资源¶
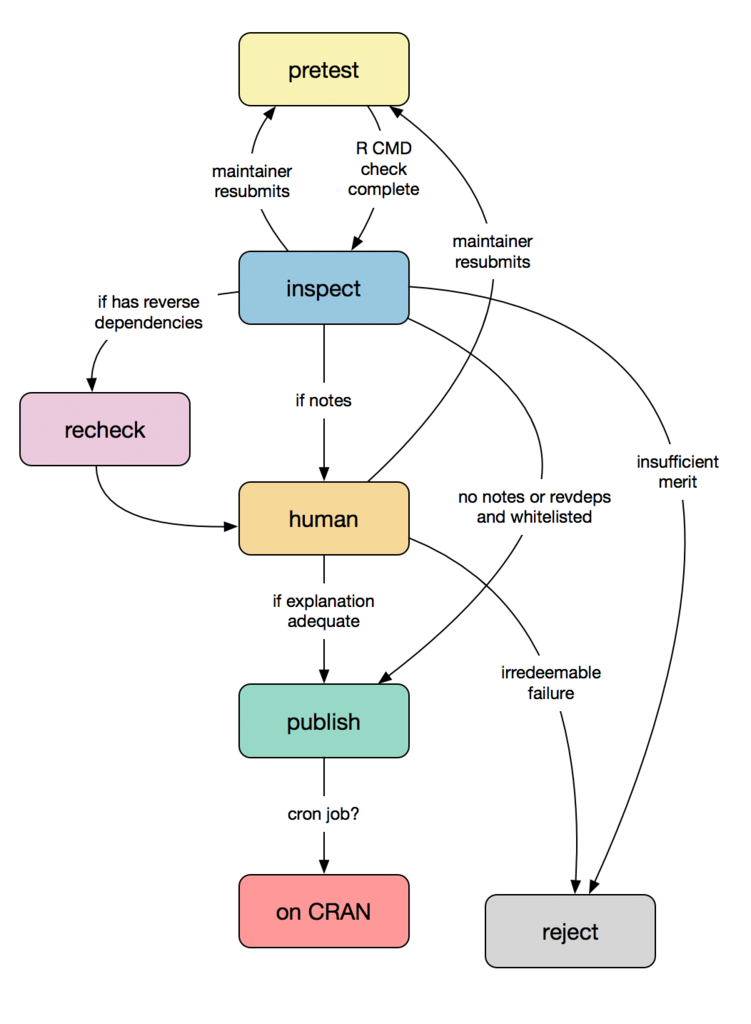
本仓库提供的资料和建议为你提交包到CRAN上分享给全世界的人们使用做好准备。
15、课程 - 贝叶斯数据分析
一些贝叶斯数据分析的学习资料。
- 相关资料链接: https://avehtari.github.io/BDA_course_Aalto/index.html
- Aki Vehtari: https://users.aalto.fi/~ave/
- Bayesian Data Analysis: https://stat.columbia.edu/~gelman/book/
- 仓库: https://github.com/avehtari/BDA_course_Aalto
历史上的本周¶
贡献者(GitHub ID)¶
「Openbiox 生信周刊」运维小队:
@ShixiangWang(王诗翔)@kkjtmac(阚科佳)@NiEntropy(赵启祥)@He-Kai-fly(何凯)@JnanZhang(张佳楠)@Tomcxf(陈啸枫)@wangdepin(王德品)
订阅¶
这个周刊每周日发布,同步更新在微信公众号「优雅R」(elegant-r)上。
微信搜索“优雅R”或者扫描二维码,即可订阅。

(完)