生信爱好者周刊(第 120 期):技术写作的首要诀窍¶
这里记录每周值得分享的生信相关内容,周日发布。
本杂志开源(GitHub: openbiox/weekly),欢迎提交 issue,投稿或推荐生信相关内容。
封面图¶
 来自:https://www.decodingbio.com/p/biobyte-072-dna-printing-edible-neuromodulatory
来自:https://www.decodingbio.com/p/biobyte-072-dna-printing-edible-neuromodulatory
本周话题:技术写作的首要诀窍¶
@kkjtmac 本周的讨论话题来自科技爱好者周刊(第 288 期),里面提及了关于技术写作的一些让人启发建议:1.技术写作的评价标准是只需要把问题说清楚 2.把问题说清楚的关键,在于你的思想是否清楚。3.每篇文章保持单线结构,坚持做到一篇文章只讲一点,而且争取把这一点讲透。
生信研究¶
1、Nature Communications | 空间转录组学新方法FISHnCHIPs可准确映射细胞类型

Nature Communications上发表了名为FISHnCHIPs的空间转录组学新方法,该方法通过同时成像多个共表达基因,提高了对细胞类型和基因表达程序进行原位分析的灵敏度,比传统单基因FISH技术高出2-20倍。FISHnCHIPs能够快速、大规模地分析细胞类型,揭示细胞间相互作用的复杂性,对理解复杂组织和细胞群体具有重要意义。
- 论文链接:https://www.nature.com/articles/s41467-024-46669-y
2、Cell Research | 人工智能驱动的CAR-T治疗设计平台: CAR-Toner
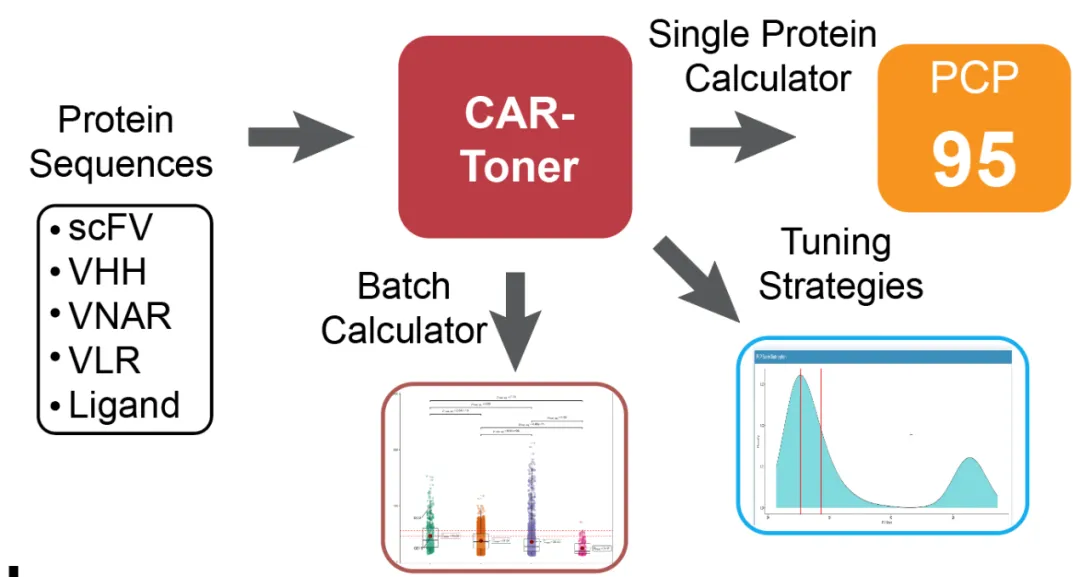
CAR-Toner取名自CAR-Tonic Signal Tuner,意为CAR-T基底信号调节器,通过人工智能深度学习技术,集齐了CAR-T设计三大功能:1. 输入单条CAR的抗原识别序列后可以一键计算反应其基底信号强度的PCP指数;2. 可同时成组输入多条CAR的抗原识别序列,一键计算各自序列的PCP指数;3. 在之前计算得出的PCP指数基础上一键给出PCP优化后的CAR序列供研究者参考(图2)。该工具界面简洁友好,对CAR-T研发者完全实现了傻瓜式一键操作功能。
- 论文链接:https://www.nature.com/articles/s41422-024-00936-1

Nature上发表的这篇综述《The status of the human gene catalogue》回顾了人类基因组注释的进展和挑战。文章指出,自人类基因组计划以来,我们对基因组的理解已从简单的基因集合转变为包含替代转录本、非编码RNA和调控元件的复杂网络。尽管对蛋白质编码基因的注释取得了进展,但非编码RNA的功能和注释仍是挑战。基因数量的估计已从最初的10万降至不到2万。文章强调了为临床使用制定通用注释标准的重要性,并提到了长读长测序技术在完善基因目录中的作用。准确的基因注释对遗传病的诊断和治疗至关重要,而新技术的发展正在帮助我们更深入地理解基因调控和基因组的复杂性。
- 论文链接:https://www.nature.com/articles/s41586-023-06490-x
4、Cell | 基于翻译后修饰的泛癌分析揭示不同癌症类型的蛋白质调控共同模式
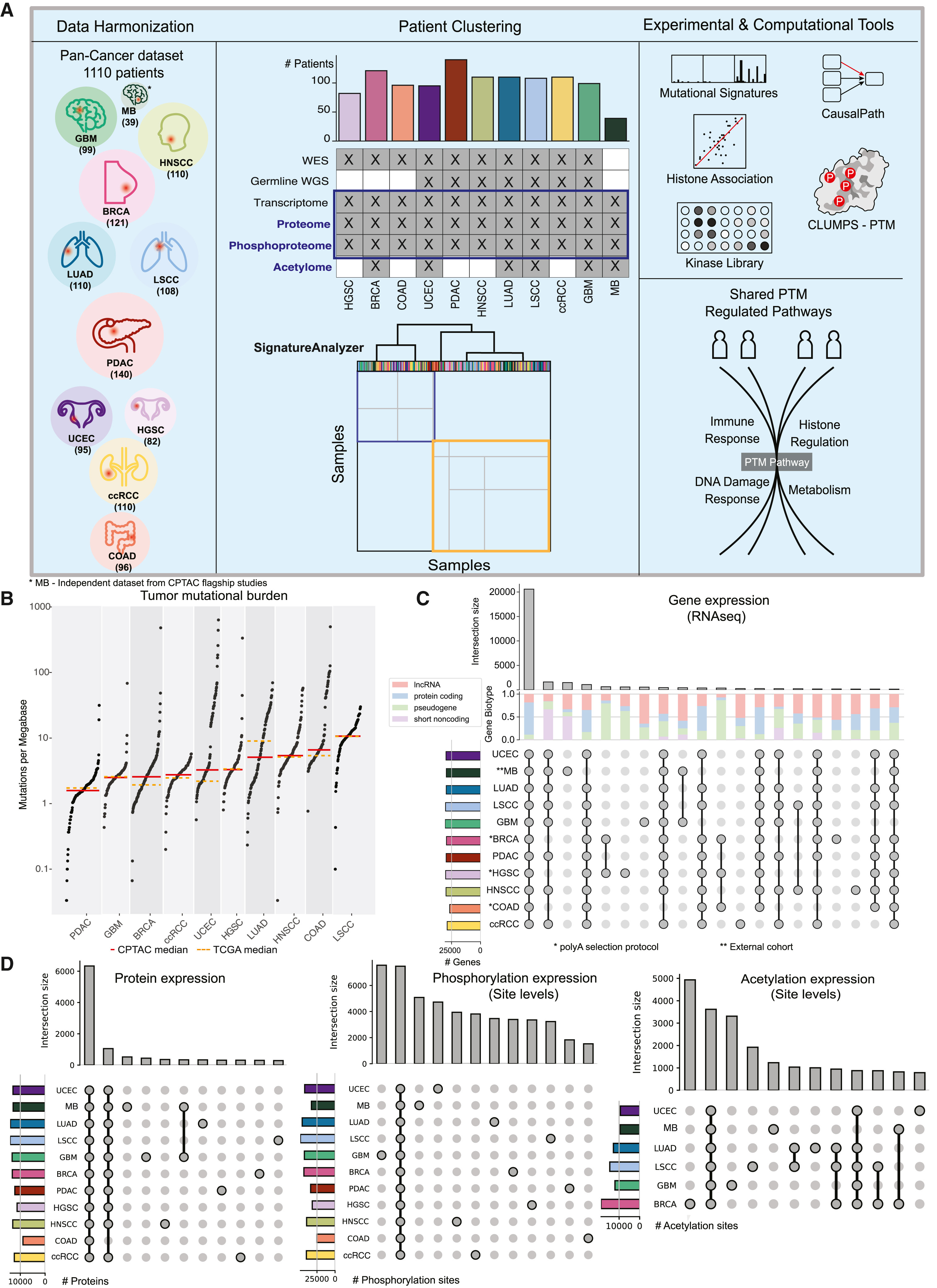
美国麻省理工学院和哈佛大学Broad研究所的研究团队在《Cell》杂志上发表了一项泛癌分析研究,揭示了不同癌症类型中蛋白质翻译后修饰(PTM)的共同调控模式。研究利用了1,110名未接受治疗的患者的样本,包括基因组、蛋白质组学和PTM数据,发现癌症进展过程中蛋白质乙酰化和磷酸化的变化模式,并识别出肿瘤亚群。研究指出,PTM参与调控多种生物学活动,并可能指示潜在的治疗途径,为癌症的个性化治疗提供了新的视角。
- 论文链接:https://www.cell.com/cell/fulltext/S0092-8674(23)00781-X#secsectitle0010
博文资讯¶
 - 工具链接:https://gitlab.com/vugene/aPEAR
- 工具链接:https://gitlab.com/vugene/aPEAR
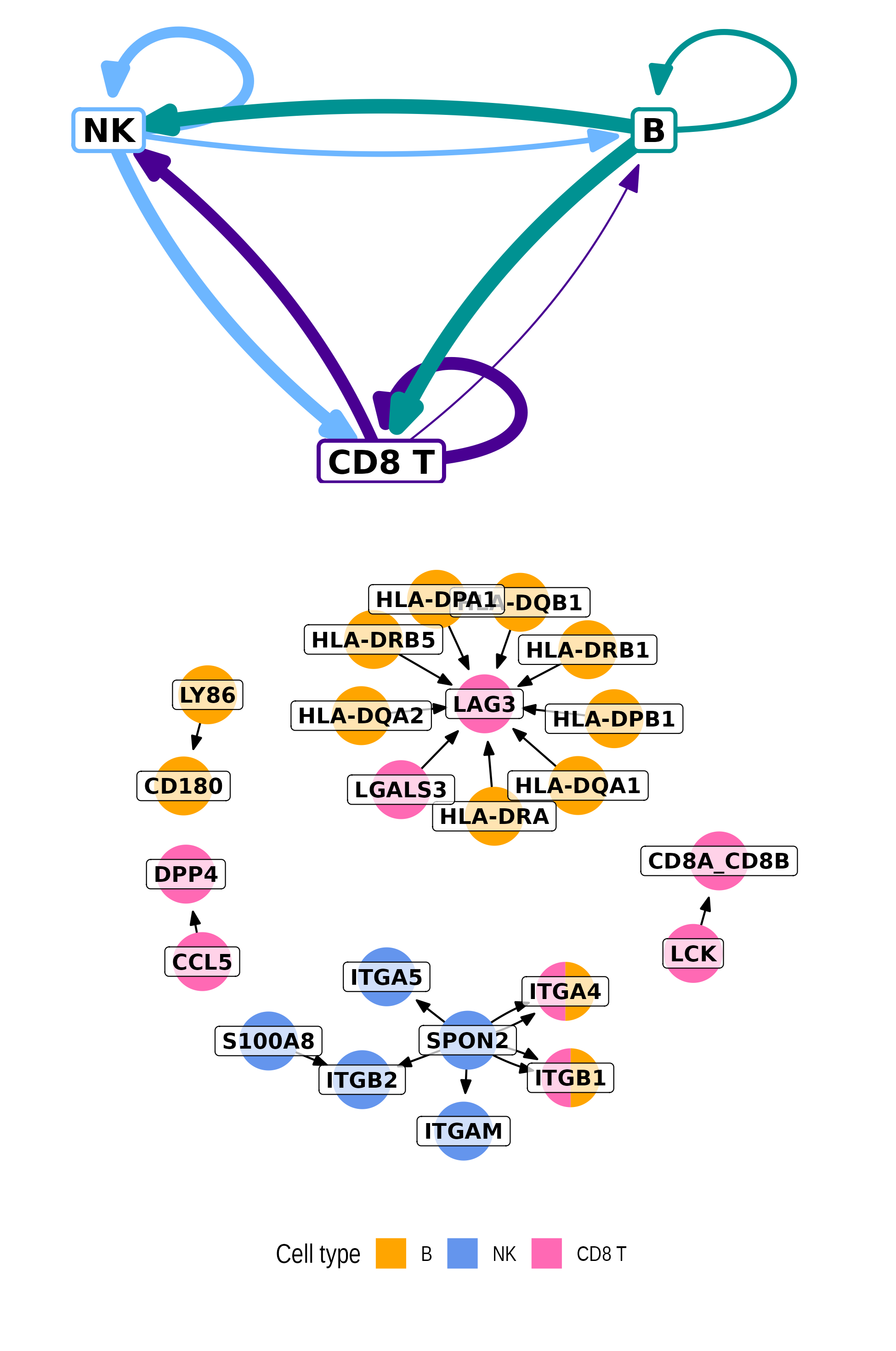
CCPlotR是一个用于可视化单细胞RNA测序数据中预测细胞间相互作用的R包。它支持多种工具生成的结果,如Liana、CellPhoneDB、NATMI等,只需要输入包含源细胞、目标细胞、配体、受体和分数的DataFrame。该包提供了六种类型的图表生成函数:cc_heatmap、cc_dotplot、cc_network、cc_circos、cc_arrow和cc_sigmoid,每种都可用于展示细胞间相互作用的不同视图。此外,该包还附带示例数据集,以帮助用户理解输入数据的格式。
- 工具链接:https://github.com/Sarah145/CCPlotR

文章介绍了R语言ggplot2包中图层的编写技巧,包括图层对象的创建、ggplot对象的渲染过程,并通过自定义图层geom_whisker的例子,展示了如何扩展ggplot2。
工具¶
8、tidygenomics | 以“tidy-way”处理基因组数据框的R包

tidygenomics是一个按tidy方式处理基因组数据框的R包,通过它可以简化基因组区间数据处理,便于整合进数据整理流程。
9、Tiny RDM | 一个更现代化的Redis桌面管理客户端
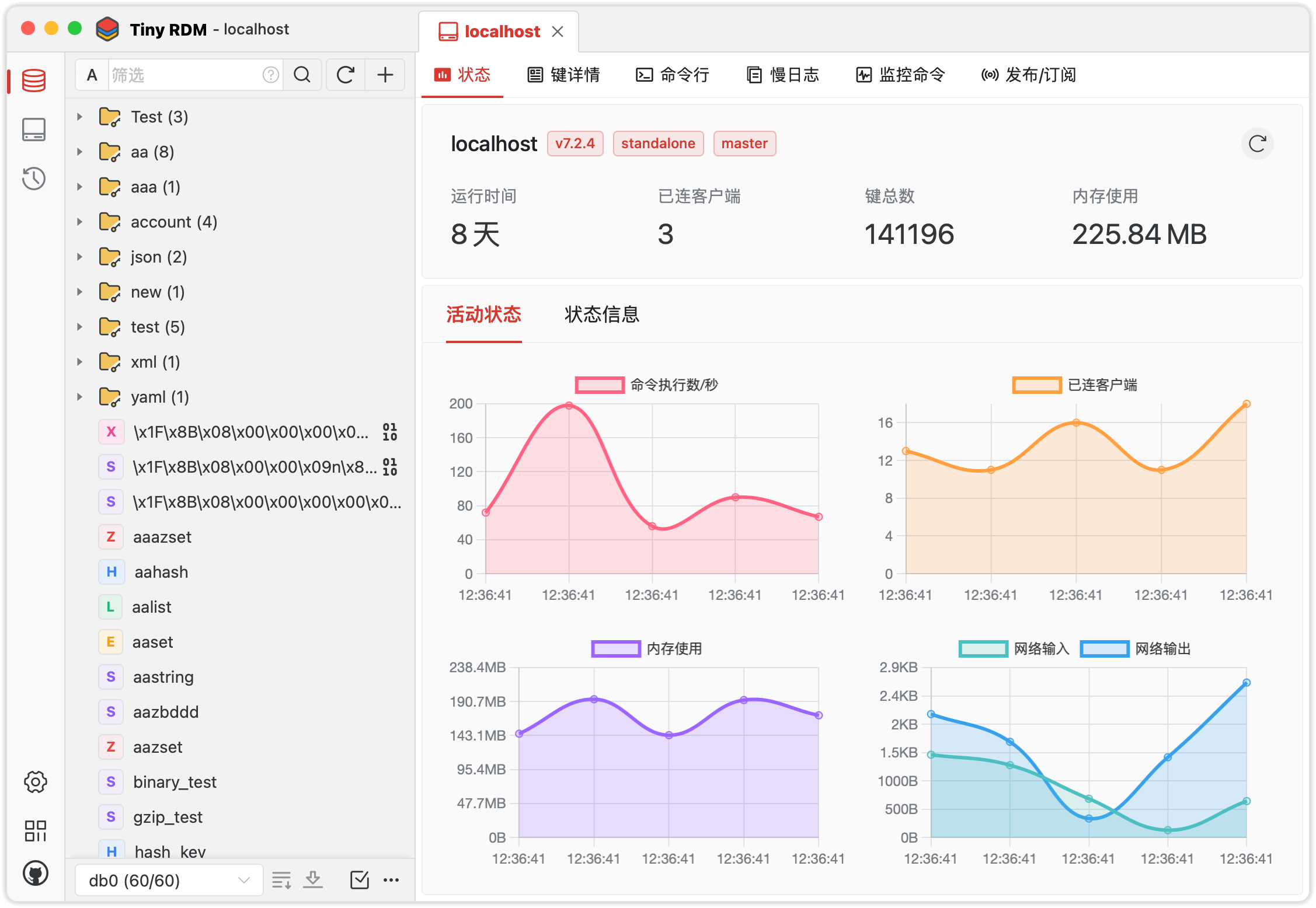
Tiny RDM(全称:Tiny Redis Desktop Manager)是一个界面现代化的轻量级Redis桌面客户端,支持Linux、Mac和Windows。
10、Chromap | 利用卷积神经网络提升纳米孔测序数据的映射与校正
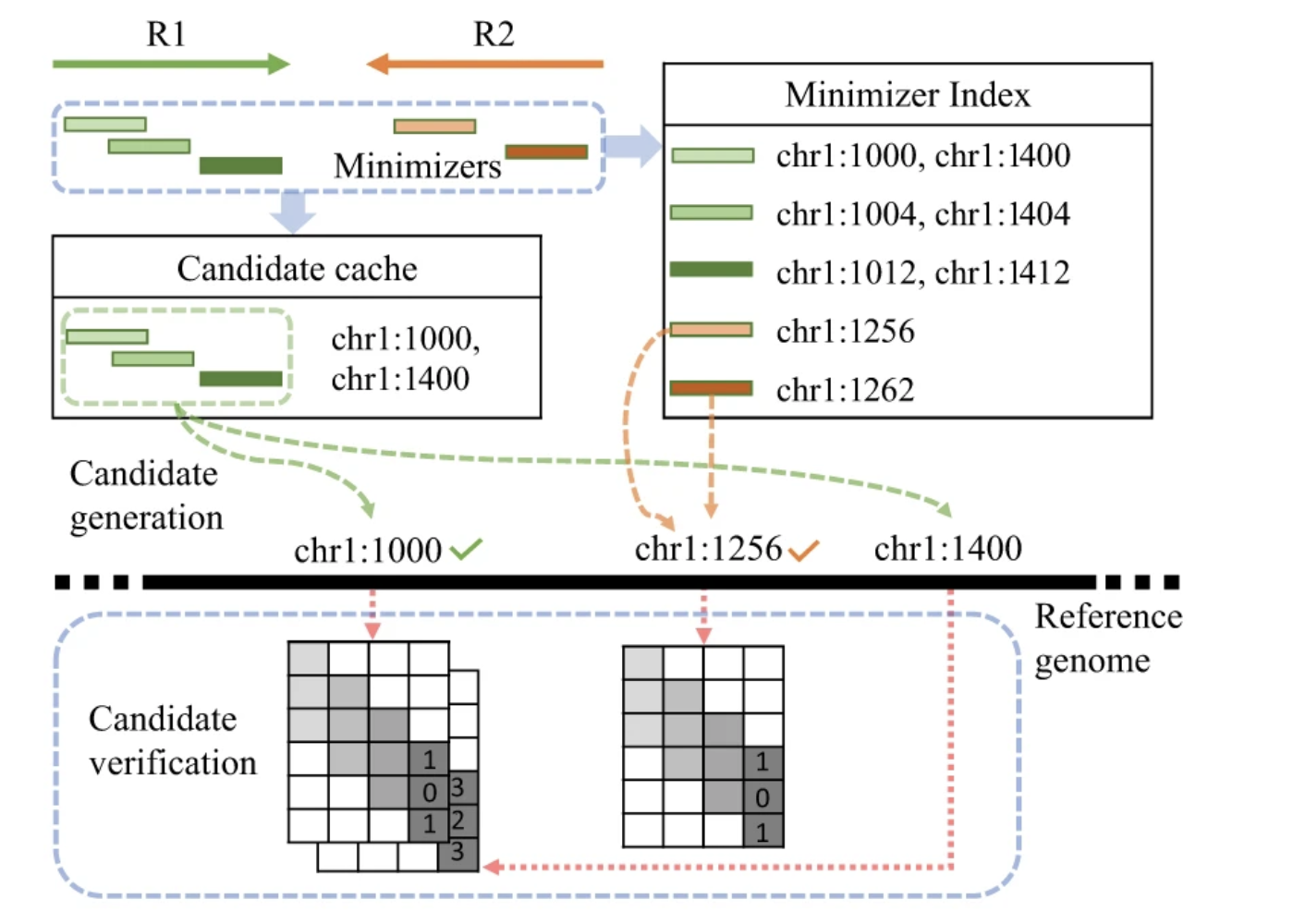
Chromap是专门设计用于处理纳米孔DNA测序数据的工具。它通过应用卷积神经网络(CNN)对长读长测序数据进行精确的序列比对和错误校正。Chromap的算法优化了映射质量,提高了错误校正的准确性,从而提升了纳米孔测序数据的可用性和可靠性。
- 论文链接:https://www.nature.com/articles/s41467-021-26865-w
- 工具链接:https://github.com/haowenz/chromap
资源¶
11、文章代码分享:孕前糖尿病导致出生缺陷的表观基因组和转录组的单细胞多模态分析
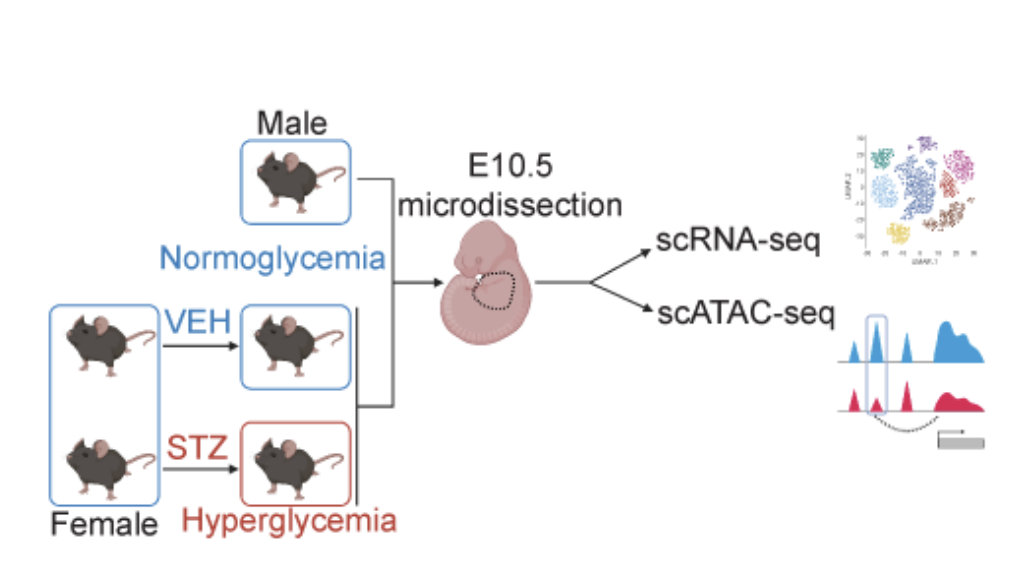
- 论文链接:https://www.nature.com/articles/s44161-023-00367-y
- 代码链接:https://github.com/SrivastavaLab-Gladstone/Nishino_DM_2022

本资源推荐了40个YouTube频道,适合生物信息学家学习新技能和知识,强调了生物信息学需要不断学习和探索。
- 资源链接:https://bioinformaticamente.com/2022/05/08/20-youtube-channels-every-bioinformatician-should-follow/amp/
历史上的本周¶
- 第79期:四千周
贡献者(GitHub ID)¶
「Openbiox 生信周刊」运维小队:
@ShixiangWang(王诗翔)@kkjtmac(阚科佳)@NiEntropy(赵启祥)@He-Kai-fly(何凯)@JnanZhang(张佳楠)@Tomcxf(陈啸枫)@wangdepin(王德品)@kongjianyang(空间阳)
订阅¶
这个周刊每周日发布,同步更新在微信公众号「优雅R」(elegant-r)上。
微信搜索“优雅R”或者扫描二维码,即可订阅。

(完)