生信爱好者周刊(第 42 期):极简主义的胜利¶
这里记录每周值得分享的生信相关内容,周日发布。
本杂志开源(GitHub: openbiox/weekly),欢迎提交 issue,投稿或推荐生信相关内容。
封面图¶

图源:2020 年黑白极简主义摄影奖-二等奖《坐下来,自由思考》,皮埃尔·佩莱格里尼
本周话题:极简主义的胜利¶
你可能听说过,极简主义(minimalism)这个词。 它指的是一种美学风格,只保留最必不可少的成分,其他都省略,追求简约。比如,极简主义的网页设计,就只有标题、正文等主要内容,其他都是空白。
kkjtmac - 除了设计中的极简主义,生信领域中也应该存在极简主义:用最简单的图说明问题,用简单的代码解决一个问题。
生信研究¶
1、Nature | 空间分辨率的染色质可及性测序新技术Spatial-ATAC-seq

2022年8月17日,樊荣教授团队和瑞典卡罗林斯卡学院Gonçalo Castelo-Branco教授团队合作利用微流控技术将组织进行空间二维编码,并与ATAC-seq技术进行结合,实现了全基因组尺度的染色质可及性测序分析,优化以后的Spatial-ATAC-seq测序结果与scATAC-seq的数据比较,质量达到相同水平,是空间表观遗传分析技术的有一个新的突破。
论文链接:https://www.nature.com/articles/s41586-022-05094-1
2、Nature Biotechnology | 解释临床蛋白质组学数据的知识图谱
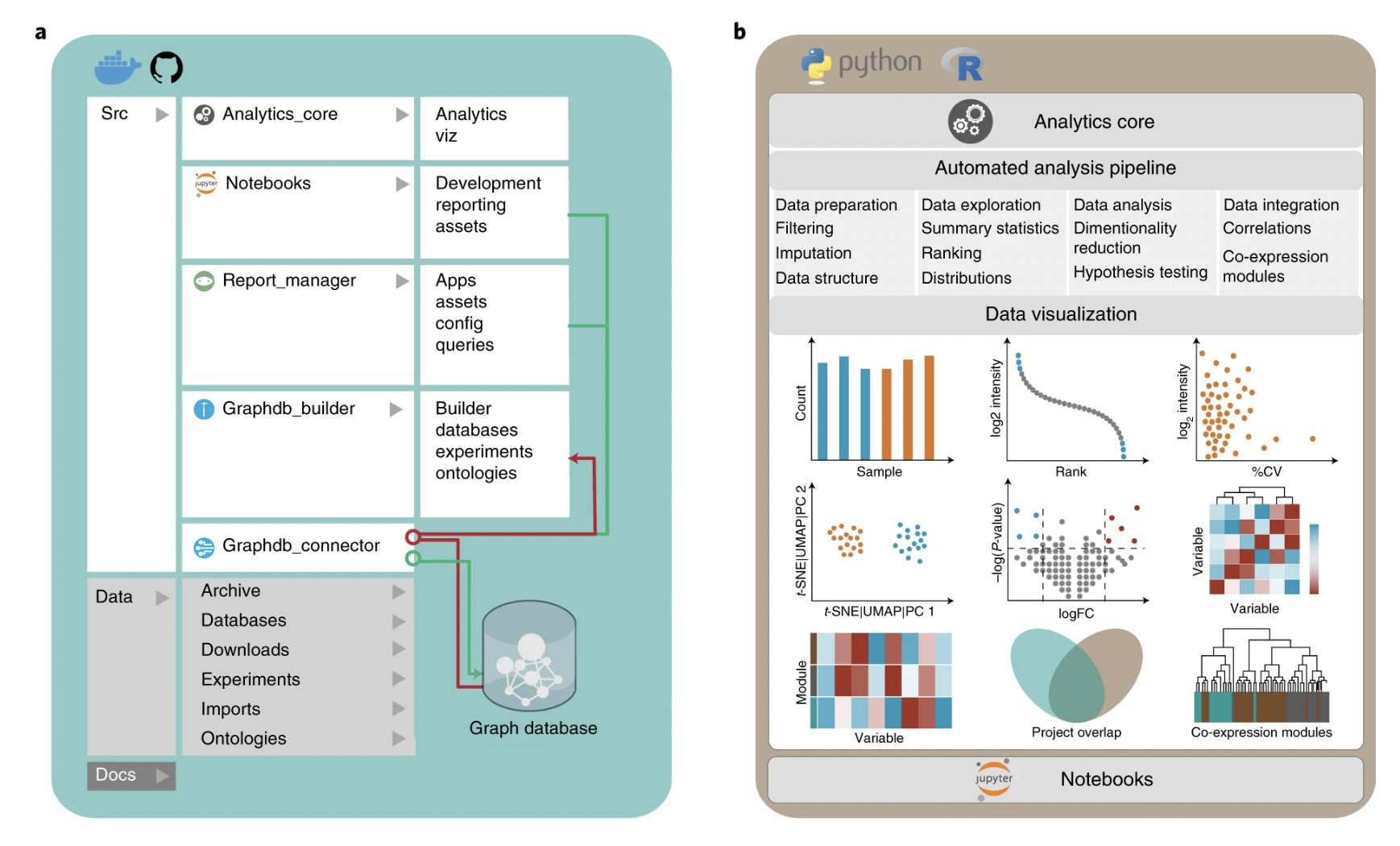
Nature Biotechnology上的这篇文章构建了一个有助于解释蛋白质组学数据的临床知识谱图 (CKG),一方面,CKG 支持具有临床意义的查询和高级统计分析,从而实现自动化数据分析、知识挖掘和可视化。
论文链接:https://www.nature.com/articles/s41587-021-01145-6

由中国科学院深圳先进技术研究院与瑞士洛桑联邦理工学院Bart Deplancke课题组、苏黎世联邦理工学院Julia Vorholt课题组合作开发的活细胞转录组测序技术(Live-seq),首次实现了让单细胞进行转录组测序后依然能够保持细胞存活。该技术兼具全基因表达分辨率和动态解析能力,是目前对单细胞转录组直接动态测量、偶联细胞现有状态和其后续表型的唯一解决方案。
论文链接:https://www.nature.com/articles/s41586-022-05046-9
博文资讯¶
4、熊说肿瘤 | ctDNA的生物学特征、检测技术问题和临床应用方向
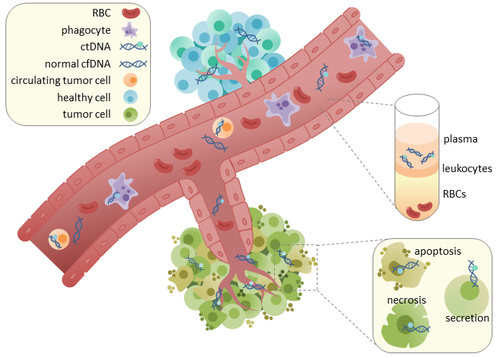
该文章结合国内外关于ctDNA专家共识和文献,对 ctDNA 目前的技术问题、应用方向和新的应用前景进行剖析,从而对ctDNA有一个相对综合的了解和把握。
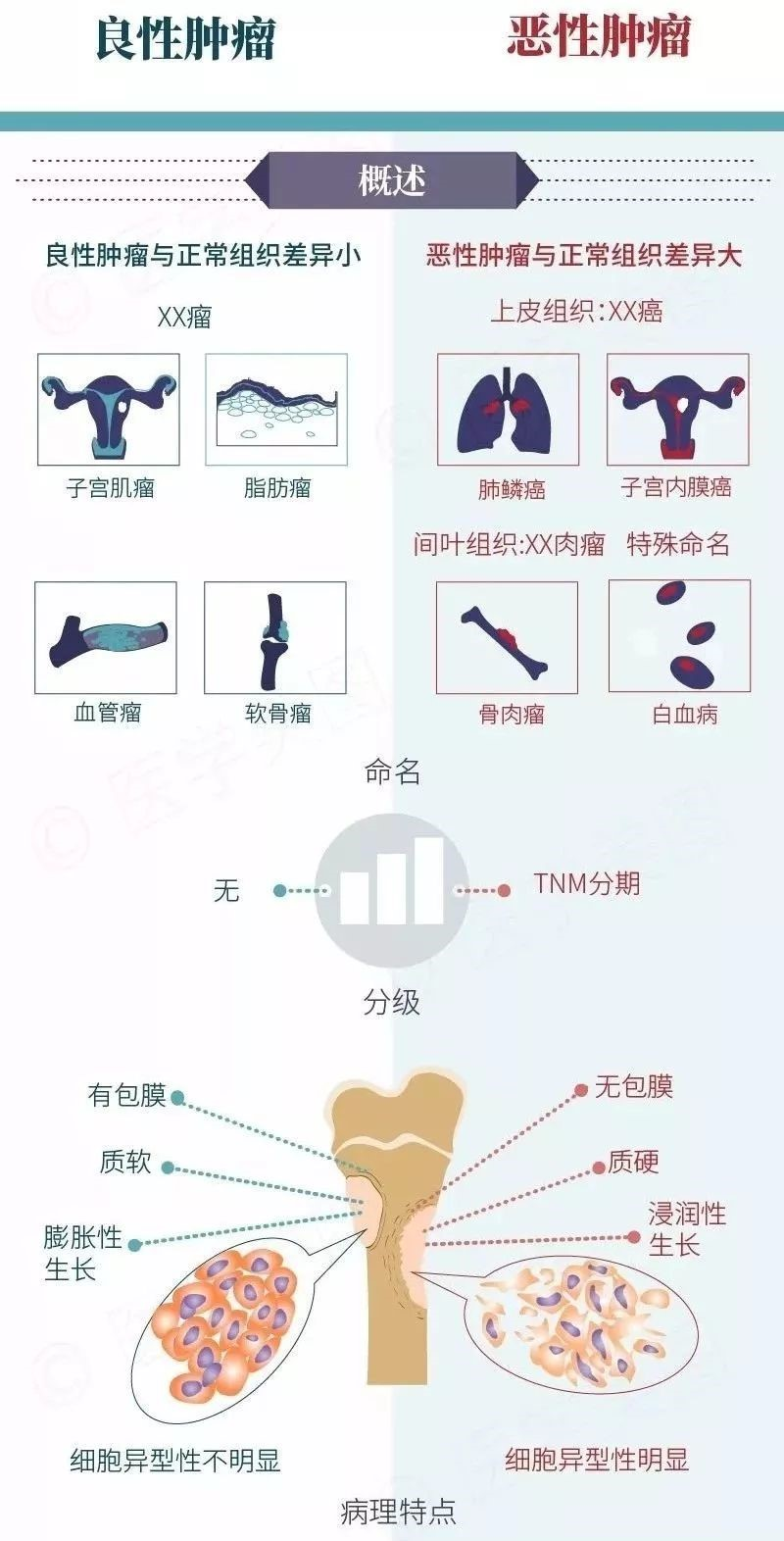
良性肿瘤,生长缓慢,与正常组织有清楚的界限;恶性肿瘤又称癌症,浸润生长,分化程度低,危害性大。对于早期或较早期癌症,大部分可以治愈,治疗时花费少、痛苦轻、时间短、疗效好。
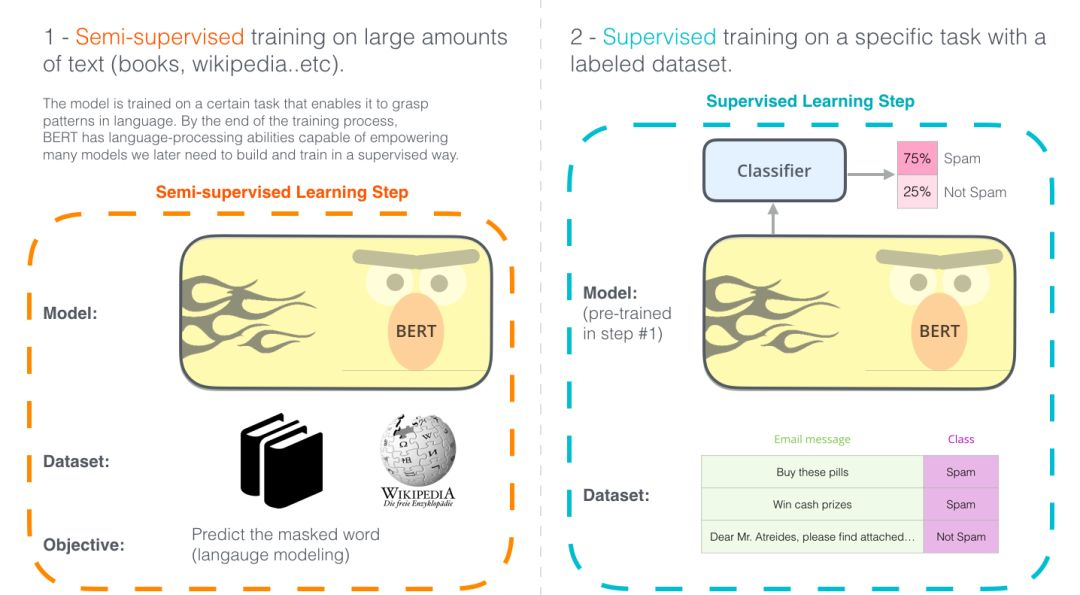
BERT是一个算法模型,它的出现打破了大量的自然语言处理任务的记录。在BERT的论文发布不久后,Google的研发团队还开放了该模型的代码,并提供了一些在大量数据集上预训练好的算法模型下载方式。Google开源这个模型,并提供预训练好的模型,这使得所有人都可以通过它来构建一个涉及NLP的算法模型,节约了大量训练语言模型所需的时间,精力,知识和资源。
(PS:实际对于门外汉来水还是不好理解哈!)
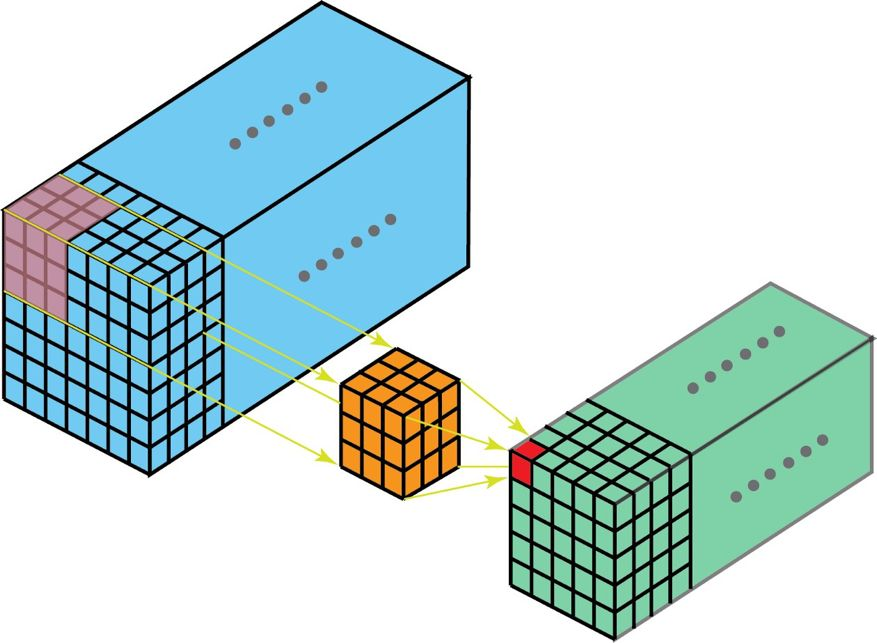
本文归纳总结深度学习中常用的几种卷积,并会试图用一种每个人都能理解的方式解释它们。
涉及如下内容: - 卷积与互相关 - 深度学习中的卷积(单通道版本,多通道版本) - 3D 卷积 - 1×1 卷积 - 卷积算术 - 转置卷积(去卷积、棋盘效应) - 扩张卷积 - 可分卷积(空间可分卷积,深度可分卷积) - 平展卷积 - 分组卷积 - 混洗分组卷积 - 逐点分组卷积
工具¶
8、scRNAtoolVis | 单细胞转录组数据的可视化展示R包
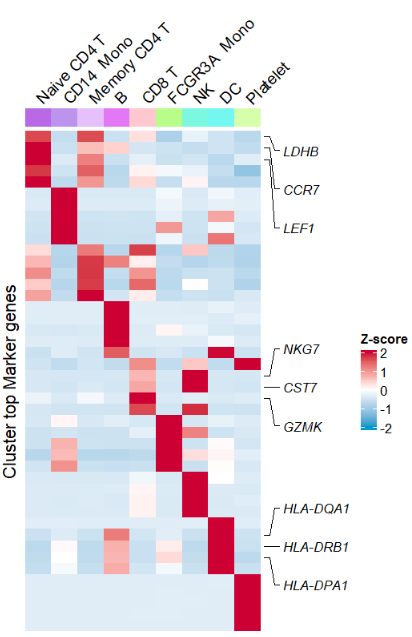
链接:https://github.com/junjunlab/scRNAtoolVis

给 HTML 文档或者 PPT 添加倒时器的 R 包「countdown」。
链接:https://github.com/gadenbuie/countdown
10、Connected Papers | 文献知识图谱构建工具
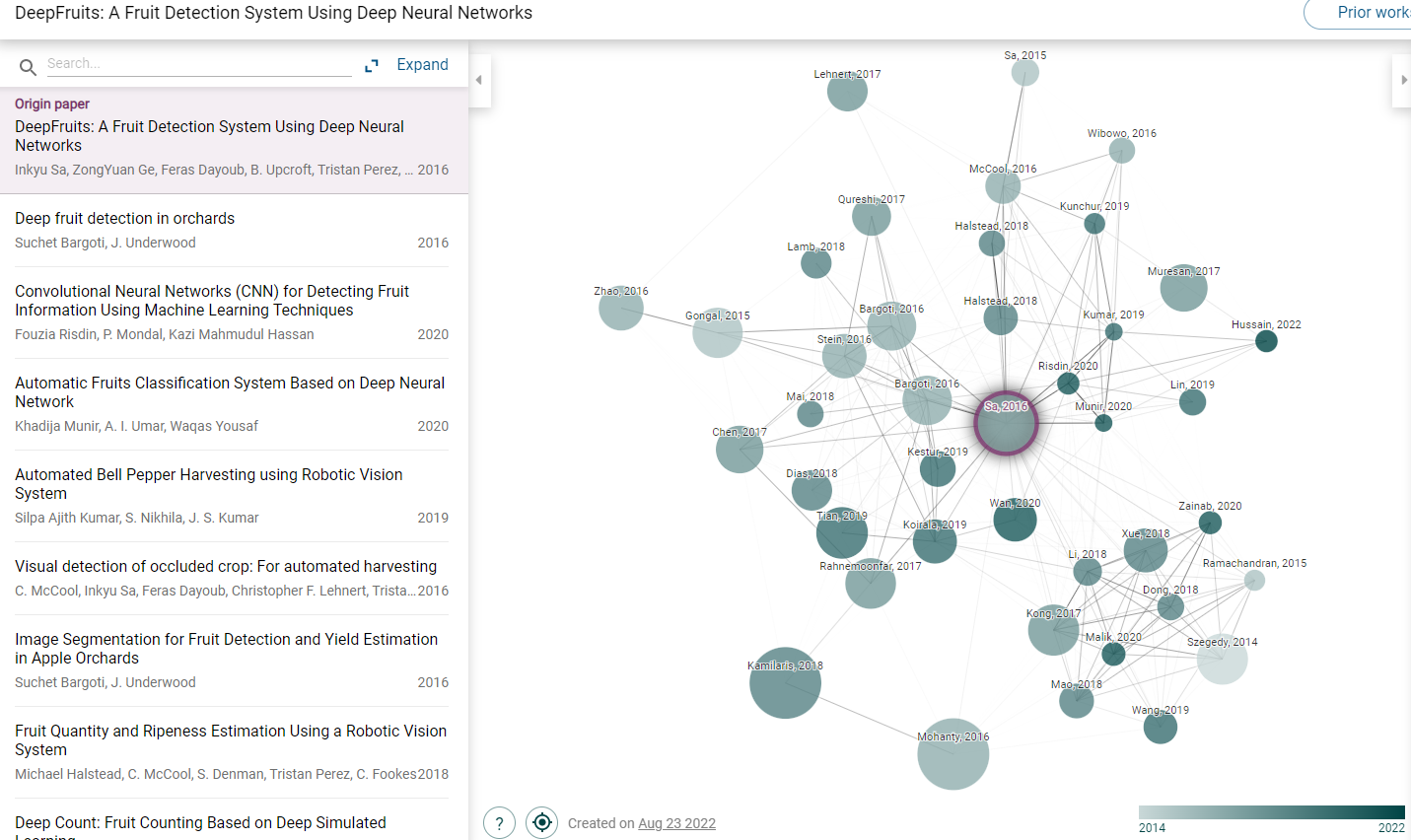
Connected Papers是一款帮助科研工作者完成文献检索和信息挖掘的可视化工具,于2020年6月正式面向大众开放,它是基于Semantic Scholar的数据构建图表,该数据库囊括了1.8亿多篇学术论文,可轻松了解某一篇文献的引用和被引用关联,分析出文献的前世今生,方便我们快速对一篇文献或者某一个领域进行调研。
链接:https://www.connectedpapers.com/
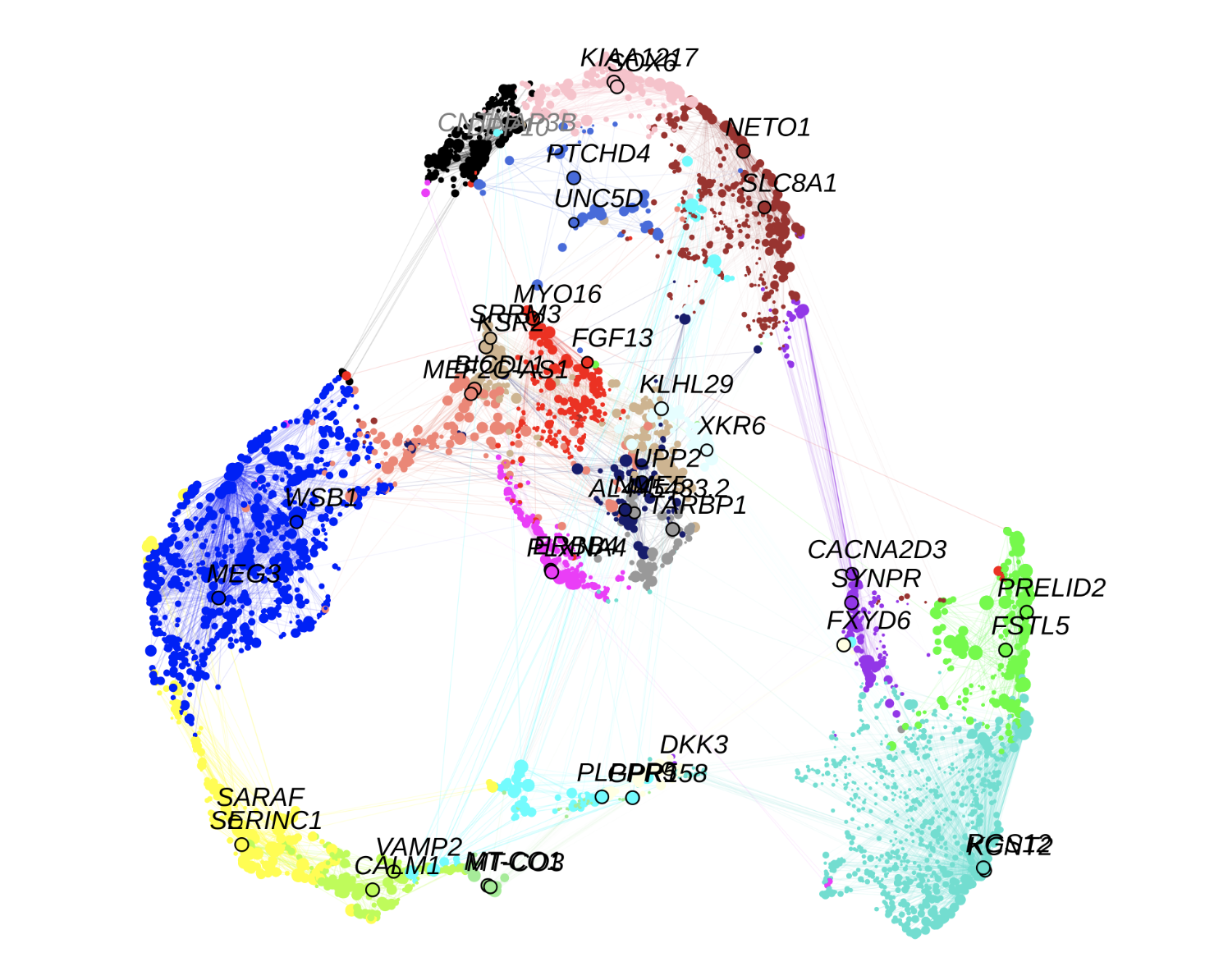
hdWGCNA,之前叫scWGCNA,提供了针对单细胞/空间转录组数据的WGCNA分析包。官方教程非常详细,从基本功能,可视化展示,富集分析,投影结果到新数据集。
链接:https://smorabit.github.io/hdWGCNA/articles/basic_tutorial.html
资源¶
12、Workshop材料:Getting Started with Shiny
本材料是为rstudio::conf 2022会议准备的,提供了初学者学习Shiny的资料(目前Shiny已经拓展到Python去了)。
在线阅读幻灯片:
- https://rstudio-conf-2022.github.io/get-started-shiny/slides/02-Day_1.html#/title-slide
- https://rstudio-conf-2022.github.io/get-started-shiny/slides/03-Day_2.html#/title-slide

单细胞转录组测序发展迅速,网上相应教程资源也都铺天盖地,笔者根据哈佛大学研究人员发布在Github上Single-cell RNA-seq analysis workshop的单细胞教程进行了整理汇总,方便大家学习!
资源链接:https://github.com/hbctraining/scRNA-seq
该资源介绍了通过R进行深度学习的相关R包和学习资料。
历史上的本周¶
- 2021:第 2 期:生信的境界与道路
贡献者(GitHub ID)¶
「Openbiox 生信周刊」运维小队:
@ShixiangWang@kkjtmac@NiEntropy@He-Kai-fly@JnanZhang@Tomcxf@wangdepin
订阅¶
这个周刊每周日发布,同步更新在微信公众号「生信协作组」(elegant-r)上。
微信搜索“生信协作组”或者扫描二维码,即可订阅。

(完)