生信爱好者周刊(第 36 期):“费钱、费力、不费脑”是中国该提倡的科研吗?¶
这里记录每周值得分享的生信相关内容,周日发布。
本杂志开源(GitHub: openbiox/weekly),欢迎提交 issue,投稿或推荐生信相关内容。
封面图¶

Xfce 桌面。via
本周话题:“费钱、费力、不费脑”是中国该提倡的科研吗?¶
本周话题来自饶毅老师的微信文章。
科学研究的目的是科学发现,经费用于发现,正常的科学是在有限的经费尽可能做更好的研究; 现在有时出现以花钱为目标,科学用于花钱,异常的科研是以有限的科研尽可能花越多的经费。 如果有中国的科研工作者提出,做“动脑少、花钱多、苦力大”的研究,也就是“费钱、费力、不费脑”,绝大多数中国纳税人应该不会同意。
大家的身边是否有类似的研究?如果看待?对目前中国此类研究的认识如何?
ShixiangWang:跟风式的研究项目个人感觉还是一种普遍的现状吧,符合中国目前总体的学术水平,是大部分研究人员在高压和完成(追求)绩效下的稳妥方案。至于是否一些项目有饶毅老师说的这么极端,仁者见仁了。
生信研究¶
1、Nature Biotechnology | 李恒团队开发不依赖于亲本的单倍型基因组组装工具hifiasm

如何不依赖亲本信息的情况下,自动进行高质量的单倍型组装是一个亟待解决的问题,哈佛大学医学院/Dana-Farber癌症研究所李恒课题组提出了一种全新的基因组组装算法hifiasm,其针对PacBio HiFi长读长测序技术和Hi-C测序技术进行了全新的设计,能够在不依赖亲本的情况下简单高效的生成高质量的单倍型组装结果,并且后续已在人类和各种不同的非人物种上证实效果良好。
论文链接:https://www.nature.com/articles/s41587-022-01261-x
2、Nature Biotechnology | CellTrek - 基于scRNA-seq和ST数据重建组织单细胞分辨率空间图谱
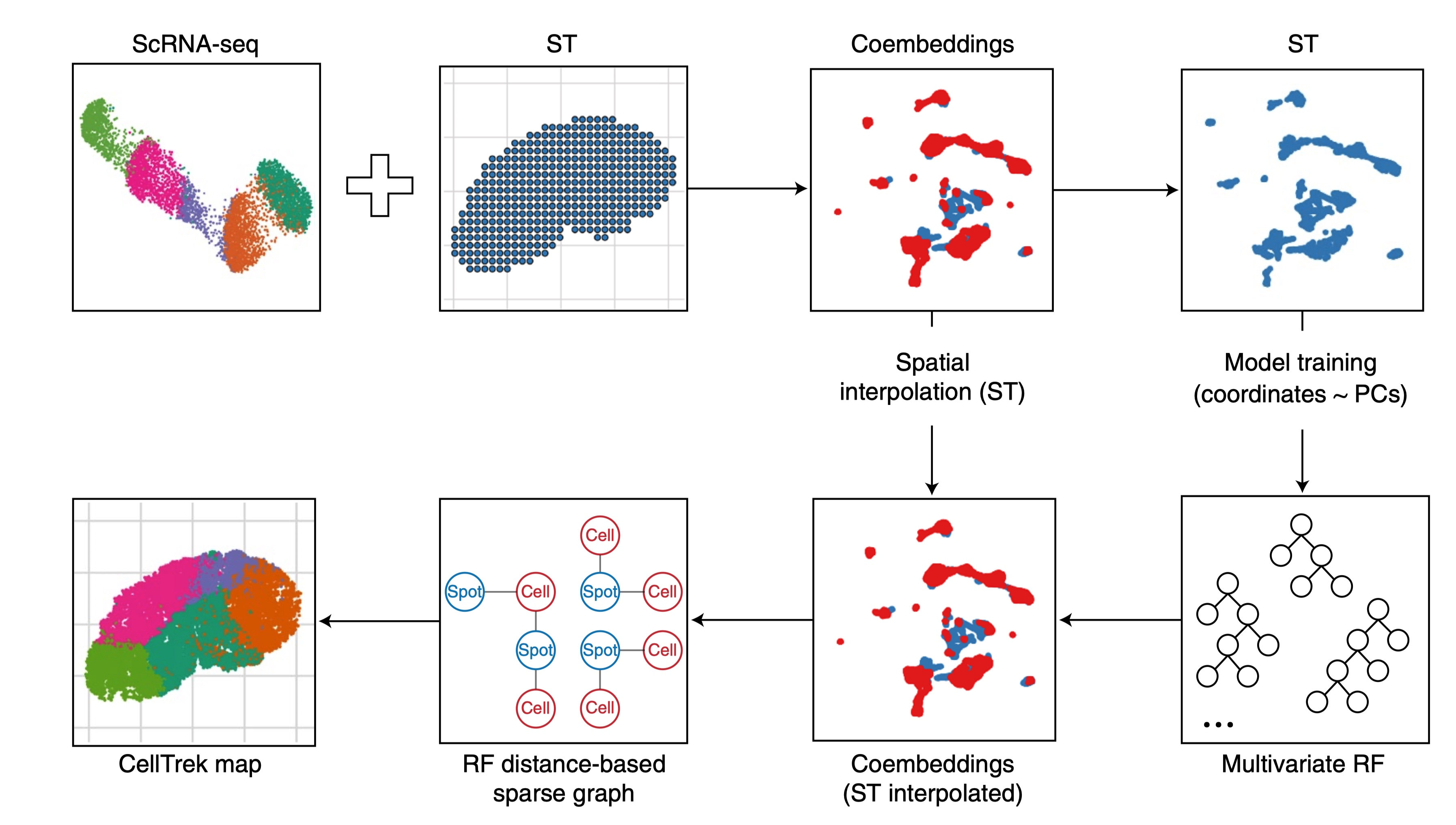
来自美国MD安德森癌症中心的团队开发了整合单细胞测序和空间转录组测序的新工具-CellTrek,该工具充分利用单细胞的高分辨率和空间转录的组织位置信息,能够进一步挖掘出组织切片中的细胞差异、空间异质性以及免疫微环境的变化。
论文链接:https://www.nature.com/articles/s41587-022-01233-1
3、Cancer Research | 全基因组倍增是人类肿瘤非整倍体变异的主要决定因素
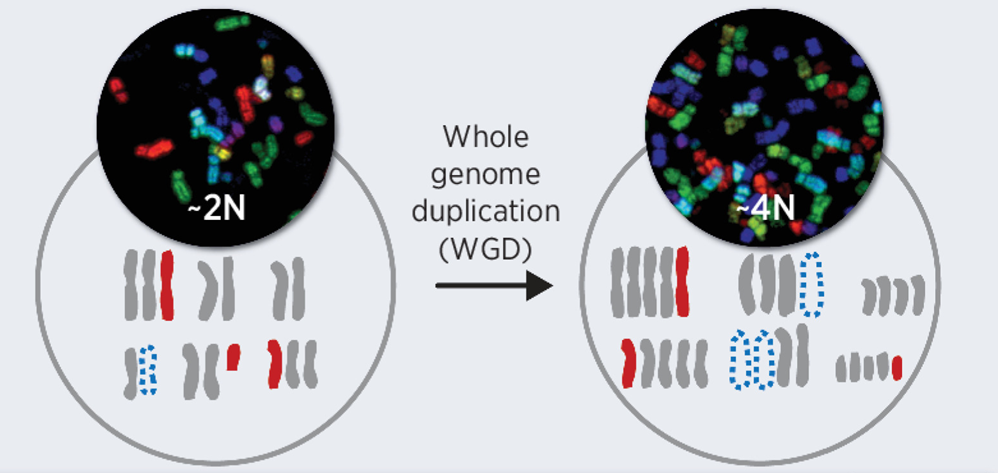
研究团队分析了来自22种肿瘤类型的5586例WGD-肿瘤和3435例WGD+肿瘤临床样本,确定了WGD-与 WGD+肿瘤中不同的非整倍体模式和染色体-臂遗传相互作用,并在人类癌细胞系中进行了验证 。结果显示,WGD塑造了人类肿瘤的非整倍体景观,是人类肿瘤非整倍体变异进化的主要决定因素。
论文链接:https://pubmed.ncbi.nlm.nih.gov/35502547/
4、Nature Machine Intelligence | 通过监督贝叶斯嵌入,对单细胞染色质可及性数据进行细胞类型注释
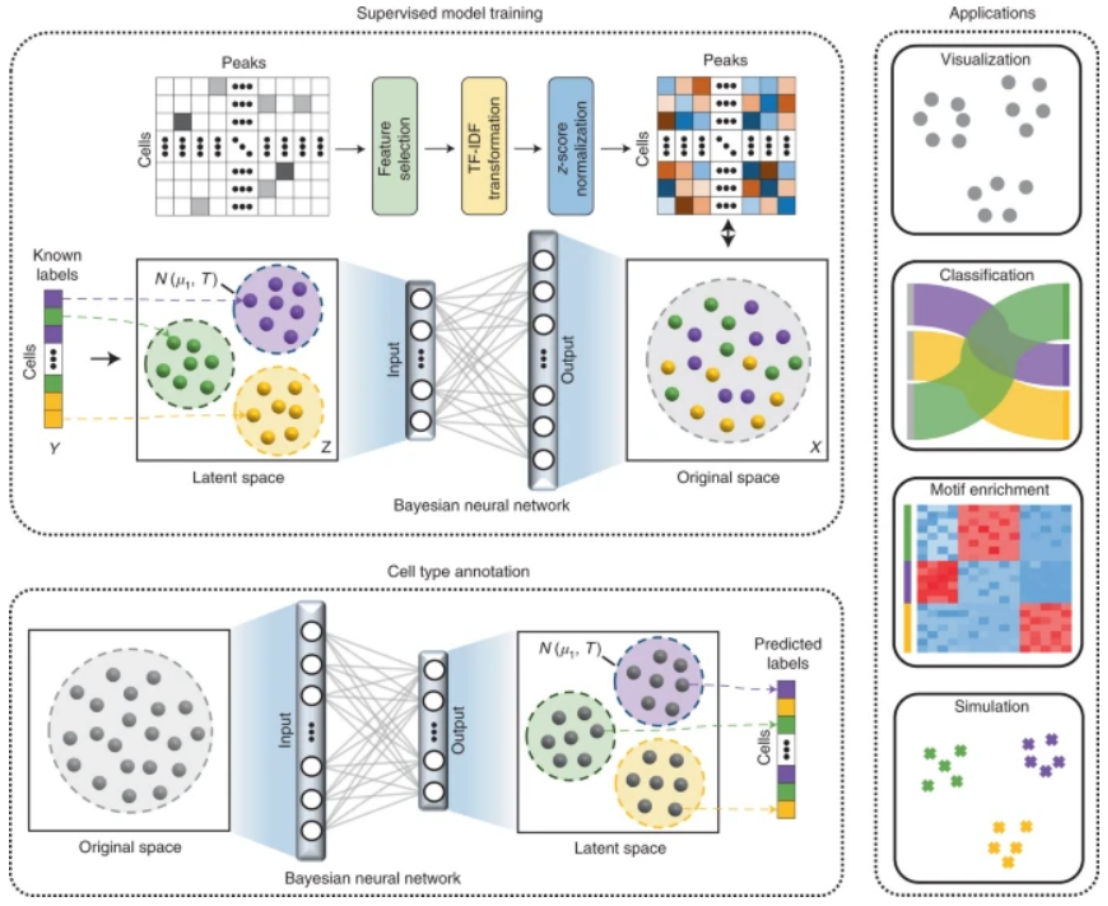
基于对多个数据集的综合实验,研究人员证明 EpiAnno 在 scCAS 数据的数据集内和跨数据集注释方面均优于基线方法。与大多数计算方法不同,经过训练的 EpiAnno 参数和学习的细胞嵌入是可解释的,并且可以通过组织特异性表达富集分析、分区遗传力分析、细胞类型特异性增强子识别和细胞类型特异性 cis-coaccessibility 分析揭示生物学见解。此外,研究人员发现 EpiAnno 不仅具有揭示细胞类型特异性基序的潜力,而且为 scCAS 数据的模拟开辟了一条新途径,这对于 scCAS 数据分析方法的发展至关重要。
论文链接:https://www.nature.com/articles/s42256-021-00432-w
博文资讯¶

发表在《英国运动医学杂志》上的一项研究发现,在晚年无法单腿站立 10 秒与未来十年内因任何原因导致的死亡风险增加近一倍有关。
原文链接:http://dx.doi.org/10.1136/bjsports-2021-105360
6、Git 不要只会 pull 和 push,试试这 5 条提高效率的命令
使用Git进行代码版本管理可谓是工程师们合作开发项目所必备技能,但实际应用中大多数人只会保存、拉取、推送等一些简单代码,遇到一些稍微复杂点的commit管理问题就束手无策,本篇推文介绍一些开发工作中较为实用的命令,助力大家解决不少疑难场景,从而提高工作效率。


本文介绍了如何免费获得微软onedrive网盘的途径以及作为同步网盘的方法。
工具¶


目前对于Python代码进行封装为exe可执行文件,见得最多的就是Pyinstaller和Nuitka,对新手而言前者更容易入手一些,但其过程体验很差,打包速度慢且最终生成的exe文件冗余庞大,而后者则将python源码转成C++,然后编译成可执行文件,使其拥有Python的开发速度,C++的运行速度,同一项目两者生成exe文件分别为3G和7M,可称为Python打包的王炸!
Github: https://github.com/Nuitka/Nuitka (含User Manual)
官网地址: https://nuitka.net/
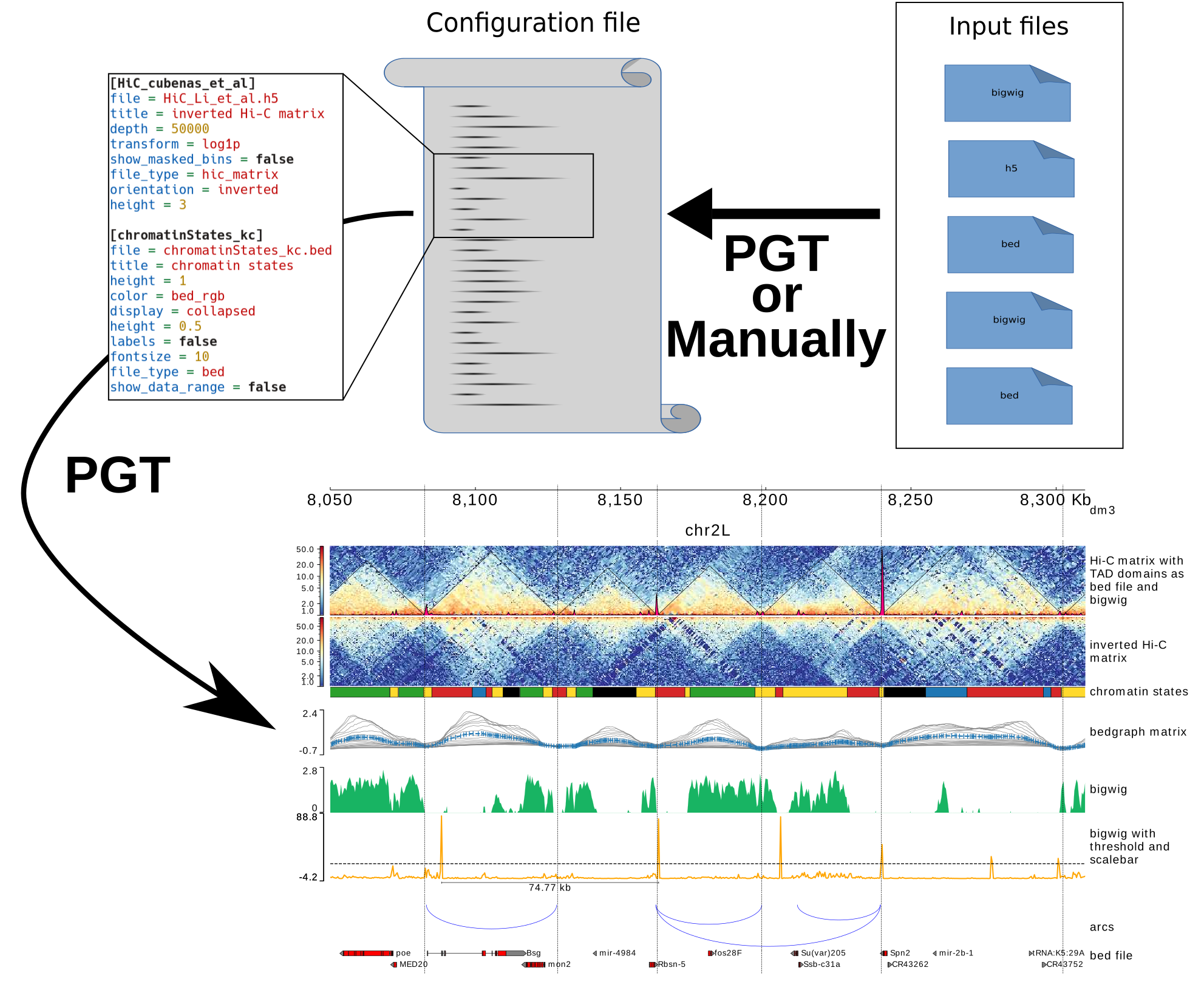
10、pyGenomeTracks - 一个基因组数据可视化的python包,可以高度定制化
11、ggdensity - 花式绘制密度图的一个gglpot2扩展包

资源¶
12、BioWebStack

一个专属生信工程师的网页,由生信重要资源汇总成的生信导航网站。常用生信数据库,在线科研绘图工具,生信杂志周刊,开源社区等等,快来探索吧!小编已经设成主页了!!!
13、Python升级:新型图像工具,玩转多维数据可视化|《自然》技术特写

napari(https://napari.org/)是一个免费、开源并且可扩展的图像查看器,适用于任意复杂(“n维”)数据,与Python生态系统紧密结合(见napari.org)。napari是三位科学家的心血结晶:加州旧金山陈·扎克伯格生物中心的显微镜专家Loïc Royer;澳大利亚墨尔本莫纳什大学从事图像分析开发的Juan Nunez-Iglesias;加州雷德伍德城的陈-扎克伯格倡议(CZI)的成像技术团队负责人Nicholas Sofroniew。
napari有类似于Adobe Photoshop的图层,允许用户叠加点、矢量、轨迹、表面、多边形、注释或其他图像。例如,研究人员可以在napari中打开一个组织的图像,点击鼠标识别细胞核,然后在Python中检索这些点,并使用它们作为细胞图像分割算法(用于识别细胞的边界)的“种子点”。然后通过将结果作为原始图像上的新图层推送到napari中,研究人员就可以评估这次分割的效果如何。
贡献者(GitHub ID)¶
「Openbiox 生信周刊」运维小队:
@ShixiangWang@kkjtmac@NiEntropy@He-Kai-fly@JnanZhang@Tomcxf@wangdepin
其他:
@shenweiyan
订阅¶
这个周刊每周日发布,同步更新在微信公众号「生信协作组」(elegant-r)上。
微信搜索“生信协作组”或者扫描二维码,即可订阅。

(完)