生信爱好者周刊(第 28 期):华大Stereo-seq系列成果揭秘超高分辨率生命全景时空图谱¶
这里记录每周值得分享的生信相关内容,周日发布。
本杂志开源(GitHub: openbiox/weekly),欢迎提交 issue,投稿或推荐生信相关内容。
Stereo-seq芯片工作原理及小鼠胚胎发育时空转录组图谱。(via)
封面图¶
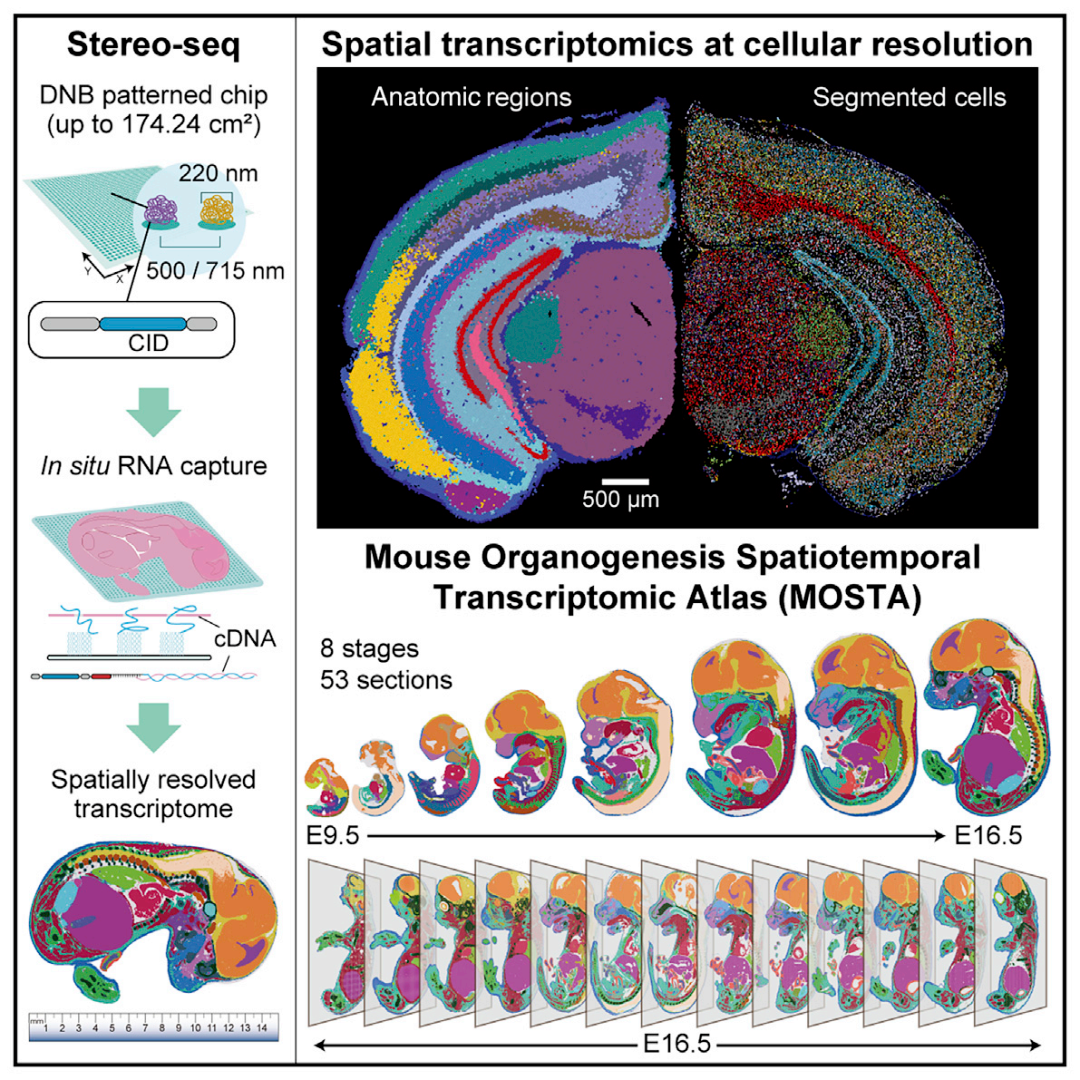
本周话题:华大Stereo-seq系列成果揭秘超高分辨率生命全景时空图谱¶
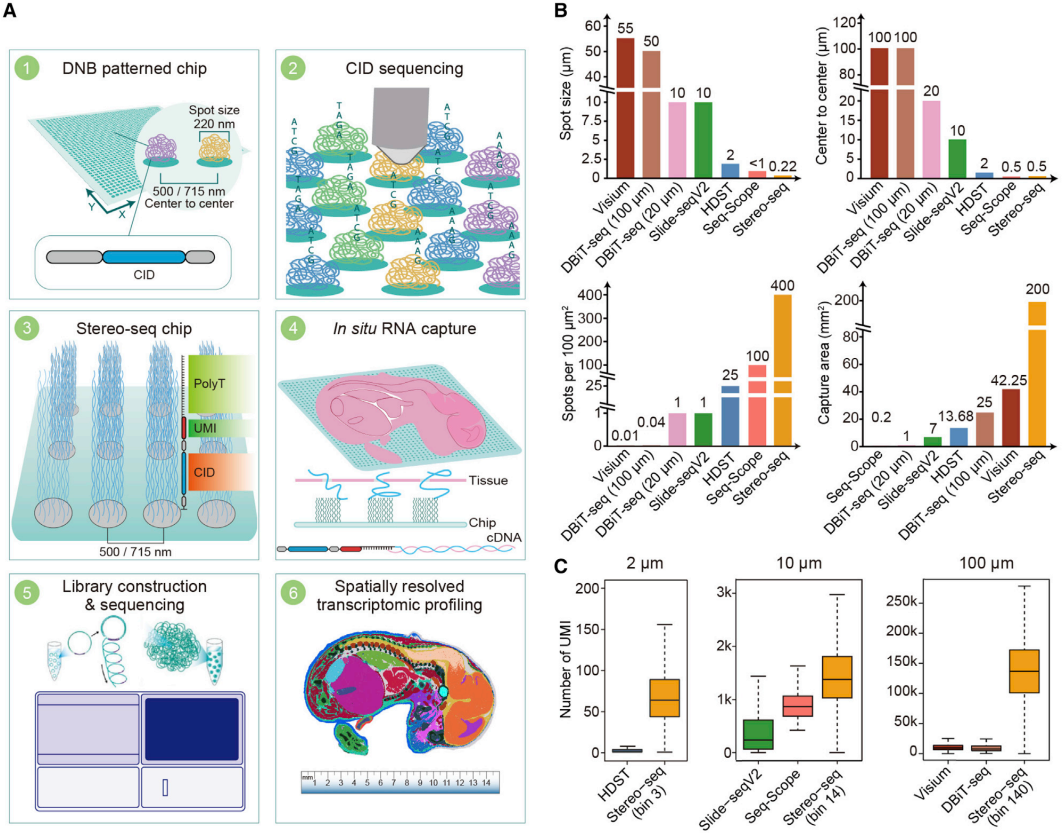
国际顶级期刊Cell和子刊Developmental Cell,上线了4篇应用Stereo-seq技术的研究文章,揭示其在小鼠、斑马鱼、果蝇、拟南芥等模式生物中的应用成果,其中Cell文章详细介绍了Stereo-seq技术原理和细节。此外,Cell出版社官网还特设专题网页(https://www.cell.com/consortium/spatiotemporal-omics),展示了以上4篇文章及4篇应用Stereo-seq技术的BioRxiv预印成果,包括食蟹猴脑时空组图谱、蝾螈脑再生时空图谱、实体瘤时空组图谱等。
相较国际同类技术,Stereo-seq通过时空捕获芯片,结合原位RNA捕获,实现了500 nm的分辨率,同时捕获面积可达13cm x 13cm,成为全球首个同时实现“纳米级分辨率”和“厘米级全景视场”的技术,且实现了基因与影像同时分析。与当前其他技术相比,在相同的精度下,Stereo-seq具备更灵敏和更强的mRNA捕获能力。该技术作为新时代的分子 “显微镜”,为重新认知器官结构、生命发育、物种演化和定义人类疾病提供了底层工具,将推动继显微镜和DNA测序技术以来的生命科学领域第三次科技革命。
生信科技动态¶
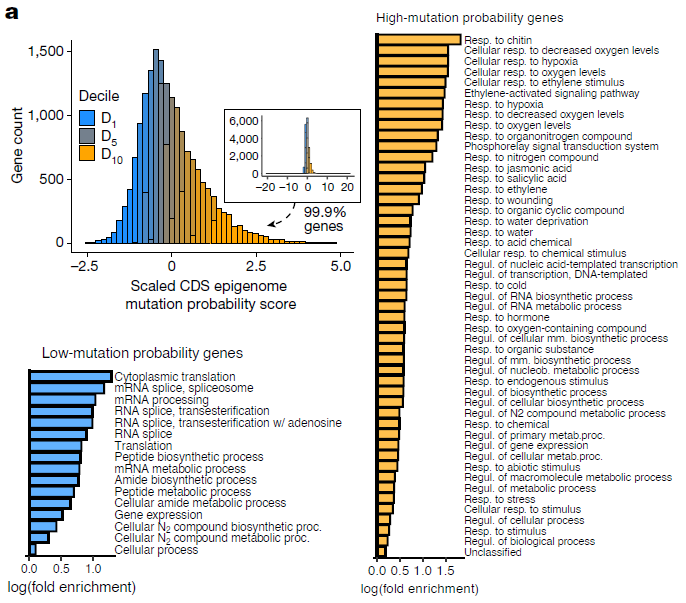
自20世纪上半叶起,突变的随机性就一直是生物演化理论的重要基础。这个观点如同生物学领域的公理,对于生物学家建立演化模型、理解遗传多样性产生了深刻影响。现在,这个经典的观点受到一项全新研究的有力挑战。
在一篇发表于《自然》杂志的论文中,一支国际研究团队通过对模式植物拟南芥的研究提出,突变的诞生不是完全随机的,相反,突变出现的区域有着明显的规律性。这个发现从根本上改变了我们对于生命演化的理解,并且有望帮助科学家培育具有更优良性状的作物,甚至帮助人类对抗癌症。
2、Nature Communication|基于2万余个肿瘤体细胞突变谱,揭示年龄对肿瘤突变数量及进化时间的影响

该研究通过分析来自TCGA、AACR GENIE和PCAWG三个项目中的数据,揭示了年龄对肿瘤中的突变数量(每年每兆碱基0.077个突变)及其进化时间的影响,绘制了具有临床意义的与年龄相关癌症体细胞突变图谱。
3、Nature | slide-DNA-seq - 自带GPS的测序技术

该研究推出的slide-seq,可以在一个直径3毫米的视野内,以10微米的分辨率,对每不同位置的聚苯乙烯珠子,添加对应空间位置的条形码,标记空间位置,之后通过PCR扩增,实现DNA原位测序。在每个阵列中,包含2万到4万个柱子,每个珠子中,可以检测到的DNA序列,约为165-421个。

文章¶
本文介绍如何使用GitHub进行版本控制与协作。
贝叶斯优化是AutoML中的重要概念,近年来变得很火热。作为一种重要的基于先验的调参/策略选择技术,贝叶斯的应用范围也很广。但这个概念对于初次接触的同学可能较难理解,经过数天的论文研读、博客/教程/代码查阅,有了这篇科普文,也手绘了一些示意图,希望尽量在一篇文章内、通俗易懂地讲清楚什么是贝叶斯优化。

工具¶
1、conflicted - An alternative conflict resolution strategy for R
R经常存在同名函数,会造成计算问题,最常见的就是filter()和select()函数。conflicted包提供了一种解决策略,一旦出现重名就报错,这样提示你一定要指定函数的包名。
library(conflicted)
library(dplyr)
filter(mtcars, cyl == 8)
#> Error: [conflicted] `filter` found in 2 packages.
#> Either pick the one you want with `::`
#> * dplyr::filter
#> * stats::filter
#> Or declare a preference with `conflict_prefer()`
#> * conflict_prefer("filter", "dplyr")
#> * conflict_prefer("filter", "stats")
2、report - bridge the gap between R’s output and the formatted results contained in your manuscript
report根据最佳实践指南(如APA的风格)自动生成模型和数据框架的报告,确保结果报告的标准化和质量。
一个处理PDF文件的命令工具。

4、gtreg - Regulatory Tables For Clinical Research with ‘gtsummary’
library(gtreg)
gtsummary::theme_gtsummary_compact()
#> Setting theme `Compact`
tbl_ae <-
df_adverse_events %>%
tbl_ae(
id_df = df_patient_characteristics,
id = patient_id,
ae = adverse_event,
soc = system_organ_class,
by = grade,
strata = trt
) %>%
modify_header(all_ae_cols() ~ "**Grade {by}**") %>%
bold_labels()
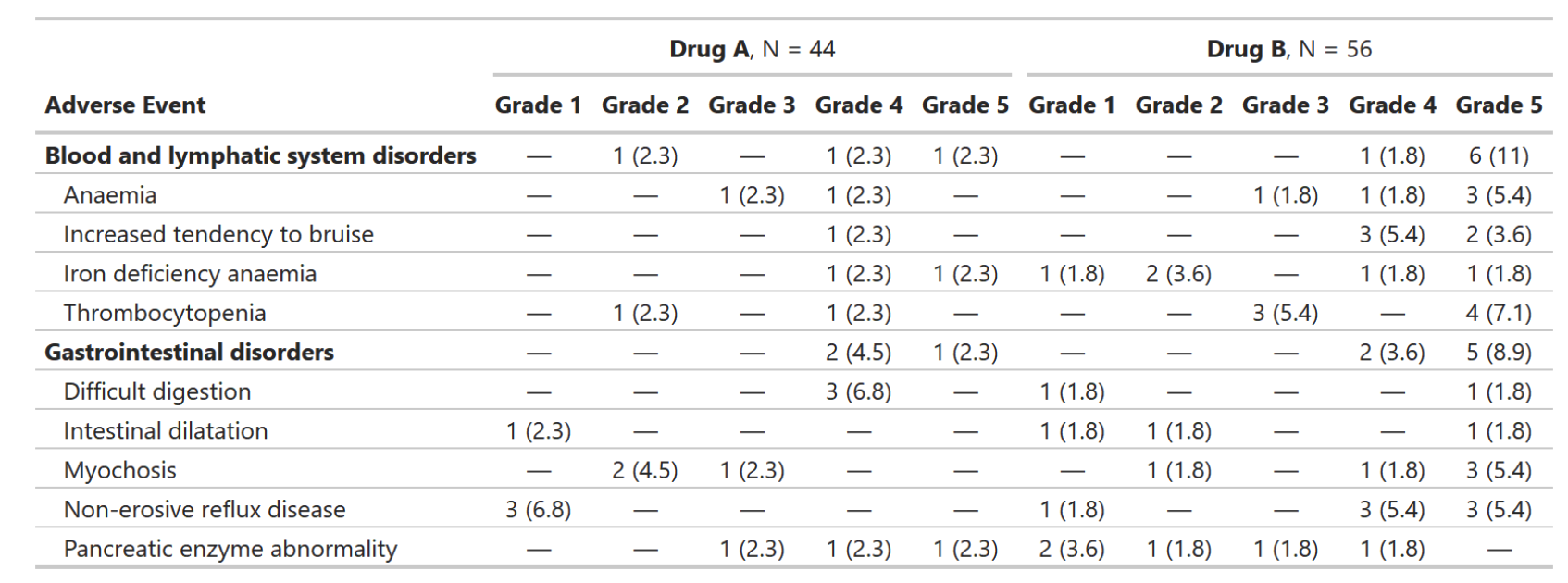
方便对R Markdown文件进行编程控制。
(rmd = parsermd::parse_rmd(system.file("minimal.Rmd", package = "parsermd")))
#> ├── YAML [4 lines]
#> ├── Heading [h1] - Setup
#> │ └── Chunk [r, 1 opt, 1 lines] - setup
#> └── Heading [h1] - Content
#> ├── Heading [h2] - R Markdown
#> │ ├── Markdown [6 lines]
#> │ ├── Chunk [r, 1 lines] - cars
#> │ └── Chunk [r, 1 lines] - <unnamed>
#> └── Heading [h2] - Including Plots
#> ├── Markdown [2 lines]
#> ├── Chunk [r, 1 opt, 1 lines] - pressure
#> └── Markdown [2 lines]
as_tibble(rmd)
#> # A tibble: 12 x 5
#> sec_h1 sec_h2 type label ast
#> <chr> <chr> <chr> <chr> <rmd_ast>
#> 1 <NA> <NA> rmd_yaml_list <NA> <yaml>
#> 2 Setup <NA> rmd_heading <NA> <heading [h1]>
#> 3 Setup <NA> rmd_chunk "setup" <chunk [r]>
#> 4 Content <NA> rmd_heading <NA> <heading [h1]>
#> 5 Content R Markdown rmd_heading <NA> <heading [h2]>
#> 6 Content R Markdown rmd_markdown <NA> <rmd_mrkd [6]>
#> 7 Content R Markdown rmd_chunk "cars" <chunk [r]>
#> 8 Content R Markdown rmd_chunk "" <chunk [r]>
#> 9 Content Including Plots rmd_heading <NA> <heading [h2]>
#> 10 Content Including Plots rmd_markdown <NA> <rmd_mrkd [2]>
#> 11 Content Including Plots rmd_chunk "pressure" <chunk [r]>
#> 12 Content Including Plots rmd_markdown <NA> <rmd_mrkd [2]>
rmd_select(rmd, by_section("Content"))
#> └── Heading [h1] - Content
#> ├── Heading [h2] - R Markdown
#> │ ├── Markdown [6 lines]
#> │ ├── Chunk [r, 1 lines] - cars
#> │ └── Chunk [r, 1 lines] - <unnamed>
#> └── Heading [h2] - Including Plots
#> ├── Markdown [2 lines]
#> ├── Chunk [r, 1 opt, 1 lines] - pressure
#> └── Markdown [2 lines]
rmd_select(rmd, by_section(c("Content", "*"))) %>%
rmd_select(has_type(c("rmd_chunk", "rmd_heading")))
#> └── Heading [h1] - Content
#> ├── Heading [h2] - R Markdown
#> │ ├── Chunk [r, 1 lines] - cars
#> │ └── Chunk [r, 1 lines] - <unnamed>
#> └── Heading [h2] - Including Plots
#> └── Chunk [r, 1 opt, 1 lines] - pressure
rmd_select(rmd, "pressure")
#> └── Chunk [r, 1 opt, 1 lines] - pressure
rmd_select(rmd, 1:3)
#> ├── YAML [4 lines]
#> └── Heading [h1] - Setup
#> └── Chunk [r, 1 opt, 1 lines] - setup
6、ggdistribute - A ggplot2 Extension for Plotting Unimodal Distributions
ggdistribute包是绘制后向或其他类型的单峰分布的扩展,这些单峰分布需要覆盖关于分布间隔的信息。它利用ggproto系统扩展ggplot2,提供额外的“geoms”、“stats”和“position”。扩展与现有的ggplot2层元素集成。
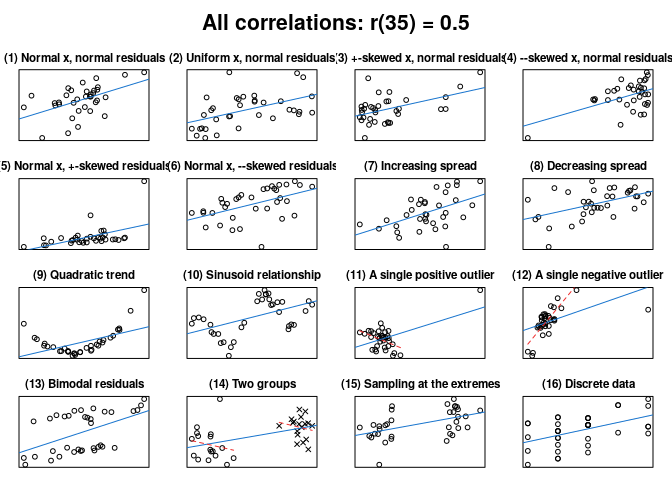
7、cannonball - 定量方法论和统计学相关函数工具
plot_r(): Draw different scatterplots with the same correlation coefficient
资源¶
赞赏¶
如果你想要支持本周刊,可以对推文进行赞赏或者提供的支付宝/微信二维码打赏。
感谢以下读者往期的赞赏:
- 李浩
订阅¶
这个周刊每周日发布,同步更新在微信公众号「生信协作组」(elegant-r)上。
微信搜索“生信协作组”或者扫描二维码,即可订阅。

(完)