生信爱好者周刊(第 25 期):从事生信工作,究竟是远见者,还是工具人?¶
这里记录每周值得分享的生信相关内容,周日发布。
本杂志开源(GitHub: openbiox/weekly),欢迎提交 issue,投稿或推荐生信相关内容。
封面图¶

本周话题:从事生信工作,究竟是远见者,还是工具人?¶
在对研究对象几乎一无所知的学科发展早期,遗传学能够帮助建立大框架,从功能上进行理解和突破;当框架基本建立完善后,生化等方法才能发挥用武之处,对细节进行修补完善;这种时间上的交错,让学科能够彼此取长补短。
相对于遗传学的框架建立能力,生物信息学其实更进一步,高通量技术的突飞猛进,促进了生信理论和技术的相应迅速发展,提供海量数据的处理方法及能力的同时,也加速了生物学研究框架建立的能力。的确,生物信息学与遗传学,或者说分子遗传学,是有更紧密的联系的。
如今,生信工作经常被人误解为跑跑流程,画画图。但其实生信的很大部分内容,是基因组学和后基因组时代的多组学,以及由这些组学所代表并蕴涵的背后生物学机制和逻辑。如今又深入到单细胞多组学,甚至亚细胞水平。加上人工智能技术的渗透和应用。生信能够成为整合这种种学科,并在独特的更全面的视角下,提供卓越远见的见地和认识,帮助其他分支学科前行。要做到这些,需要我们这些做生信的,首先不局限自己视野,别为了完成任务而完成任务,需要多思考,并尽可能回归初心,循着前人们的方向,不断追寻。
来源:公众号《不靠谱颜论》
生信科技动态¶
1、Nat. Biotechnol综述 | 纳米孔测序技术、生物信息学及应用
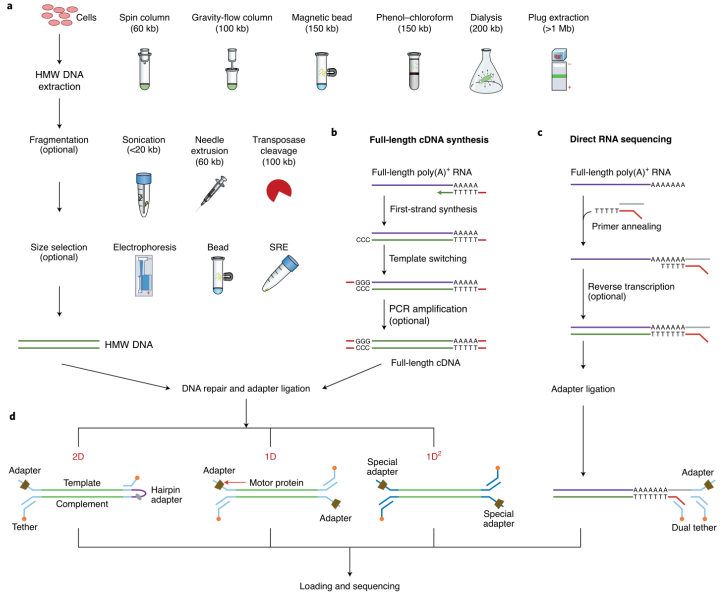
自2014年牛津纳米孔科技有限公司(Oxford Nanopore Technologies, ONT)发布第一台纳米孔测序仪MinION以来,纳米孔测序技术及其应用研究飞速增长。该技术利用纳米级蛋白质孔(纳米孔)作为生物传感器,嵌入电阻性聚合物膜中,在电解液中施加恒定电压,以产生通过纳米孔的离子电流,DNA链在马达蛋白(phi29 DNA聚合酶)的牵引下穿过纳米孔,易位期间离子电流的变化对应于传感区域中的核苷酸序列,之后使用算法进行解码,实现对单分子的实时测序。
俄亥俄州立大学Kin Fai Au团队在Nature Biotechnology发表综述文章“Nanopore sequencing technology, bioinformatics and applications”,系统介绍了纳米孔测序技术的发展,讨论了ONT数据在准确性、读长和通量方面的改进,并描述了应用于ONT数据的主要生物信息学方法以及纳米孔测序当前的主要应用。
2、Nature Protocols | 基于机器学习和并行计算的代谢组学数据处理新方法
代谢组学是对某一生物或细胞在一特定生理时期内所有代谢产物同时进行定性定量分析的学科,被广泛用于揭示小分子与生理病理效应间的关系。目前,代谢组学已经被应用于药物开发的各个阶段(如药物靶标识别、先导化合物发现、药物代谢分析、药物响应和耐药研究等)。基于代谢组学的高性价比特性,它被药学领域的研究者给予了厚望,有望加速新药开发的进程。然而,代谢组领域还面临着严重的信号处理与数据分析问题,对其在新药研发中的应用构成了巨大挑战。为了有效消除由环境、仪器和生物因素所引入的不良信号波动,就需要开发针对代谢组信号系统优化的新方法,为不同组学研究量身定制最优的数据分析策略。
该工作报道了一套基于机器学习和并行计算的优化组学信号处理策略的新方法。该方法通过大规模扫描现有的海量信号处理流程,针对用户给定的代谢组学原始数据,可以快速地优化出性能最佳的组学数据处理流程。这一方法实现了对药学领域常见的“时间序列”和“多分类”代谢组学问题的数据处理,对药物靶标发现、药物代谢、药物响应与疾病发生发展的病理学机制研究都具有重要的价值。
3、Nature Genetics | GWAS分析揭示DNA甲基化与遗传变异关联,提供基因组调控分子机制新见解
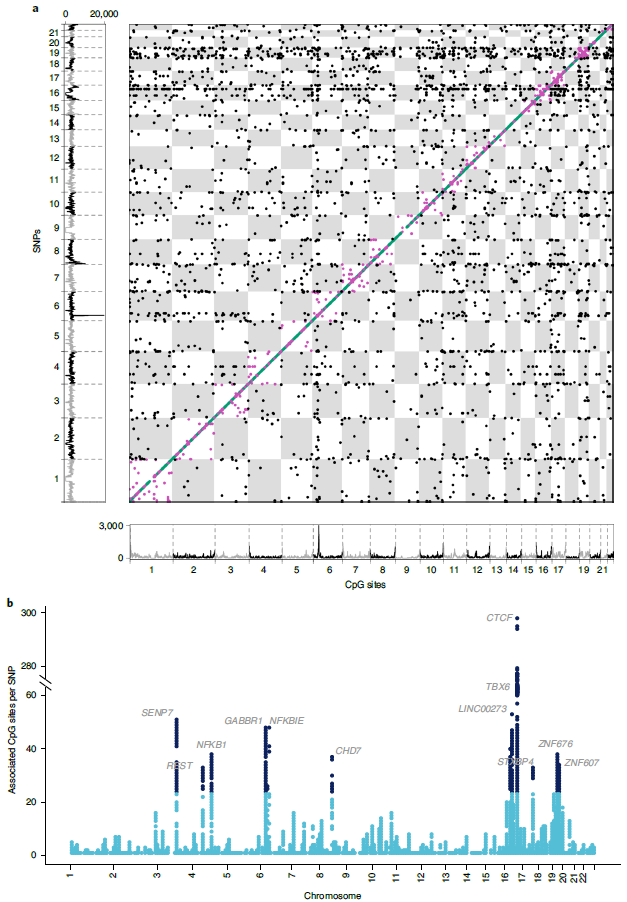
DNA甲基化在决定基因组结构和功能方面起着关键作用,包括细胞分化和基因表达的调控。已有研究证明,DNA甲基化紊乱与动脉粥样硬化、癌症、肥胖、2型糖尿病、神经精神疾病以及其他复杂的多因素疾病的发生发展有关,可作为独立因素预测全因死亡率。因此,进一步了解影响DNA甲基化的机制,有望为鉴定基因组调控因子、分子表型和疾病发展的生物学途径提供新的见解。
无论是顺式(同一染色体)还是反式(跨染色体间),DNA甲基化均可受到潜在遗传变异的影响。其中,由于反式DNA甲基化在调控基因组功能和影响多种生物过程中发挥关键的调控作用,对其有影响的遗传变异值得特别关注。本研究在之前工作的基础上,进一步解析了调控基因变异与基因表达、分子相互作用、表型变异和疾病易感性之间的分子机制,促进了人们对基因变异与人类表型之间潜在联系途径的理解。
文章¶
学习生信,总绕不开需要学习使用Linux系统。
在过去,对于重度依赖Windows系统的人来说,往往只能通过虚拟机的方式,来尝试安装和使用Linux。而现在,“适用于Linux的Windows子系统(Windows Subsystem for Linux,简称为WSL)”让这个过程变得容易许多。
2、Installing the R kernel in Jupyter Lab
对于喜欢Jupyter Lab的朋友可以试试。之前折腾过,但还是不喜欢Jupyter的编码方式,没有RStudio舒适自然。
Nginx 是一个高性能的 HTTP 和反向代理服务器,特点是占用内存少,并发能力强,事实上 Nginx 的并发能力确实在同类型的网页服务器中表现较好。
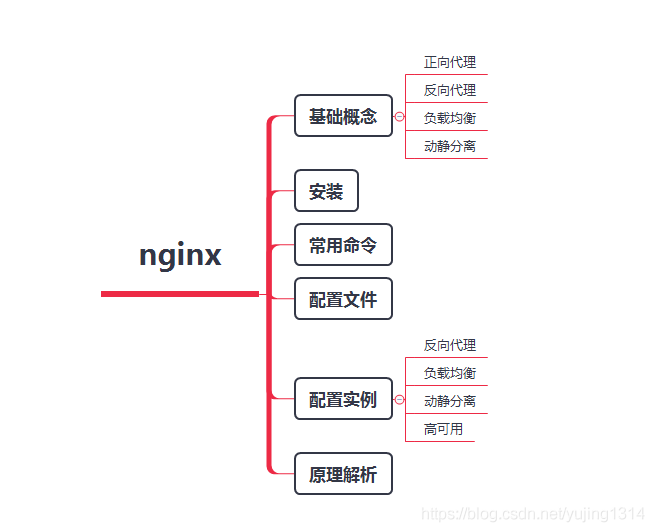
工具¶
1、skimr - A frictionless, pipeable approach to dealing with summary statistics
通过字符可视化数据汇总信息。
skim(iris)
## ── Data Summary ────────────────────────
## Values
## Name iris
## Number of rows 150
## Number of columns 5
## _______________________
## Column type frequency:
## factor 1
## numeric 4
## ________________________
## Group variables None
##
## ── Variable type: factor ───────────────────────────────────────────────────────────────────────────
## skim_variable n_missing complete_rate ordered n_unique top_counts
## 1 Species 0 1 FALSE 3 set: 50, ver: 50, vir: 50
##
## ── Variable type: numeric ──────────────────────────────────────────────────────────────────────────
## skim_variable n_missing complete_rate mean sd p0 p25 p50 p75 p100 hist
## 1 Sepal.Length 0 1 5.84 0.828 4.3 5.1 5.8 6.4 7.9 ▆▇▇▅▂
## 2 Sepal.Width 0 1 3.06 0.436 2 2.8 3 3.3 4.4 ▁▆▇▂▁
## 3 Petal.Length 0 1 3.76 1.77 1 1.6 4.35 5.1 6.9 ▇▁▆▇▂
## 4 Petal.Width 0 1 1.20 0.762 0.1 0.3 1.3 1.8 2.5 ▇▁▇▅▃
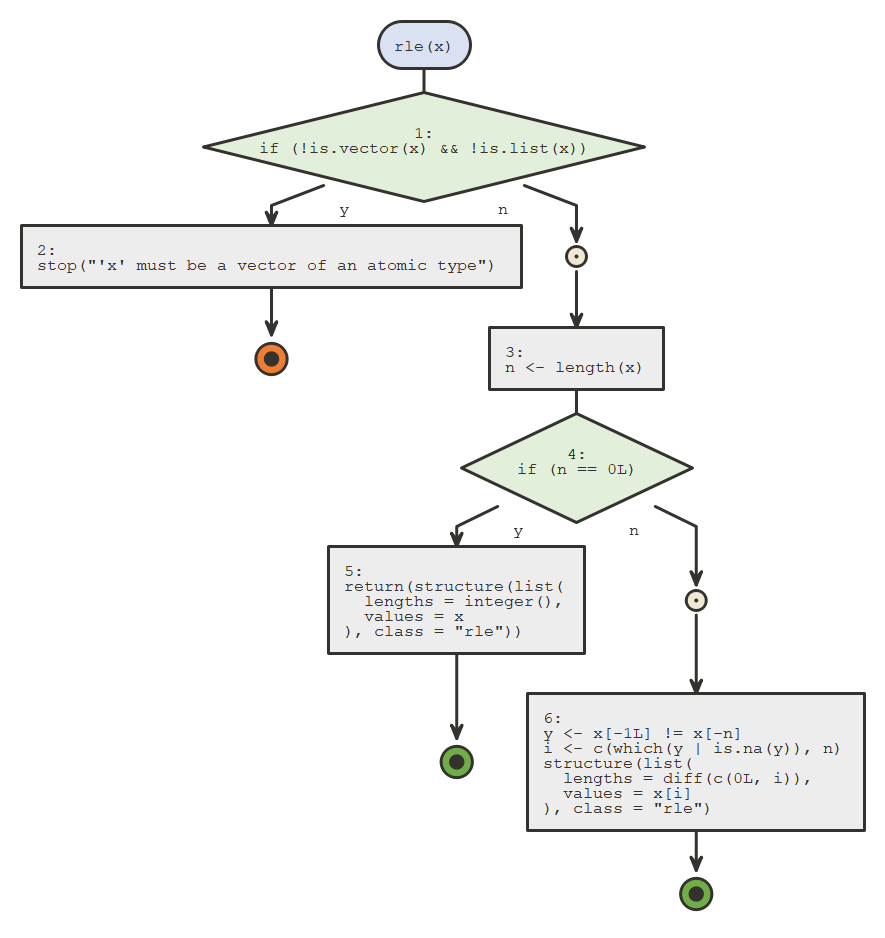
2、flow - View and Browse Code Using Flow Diagrams
输入你的R代码,它给你返回流程图。
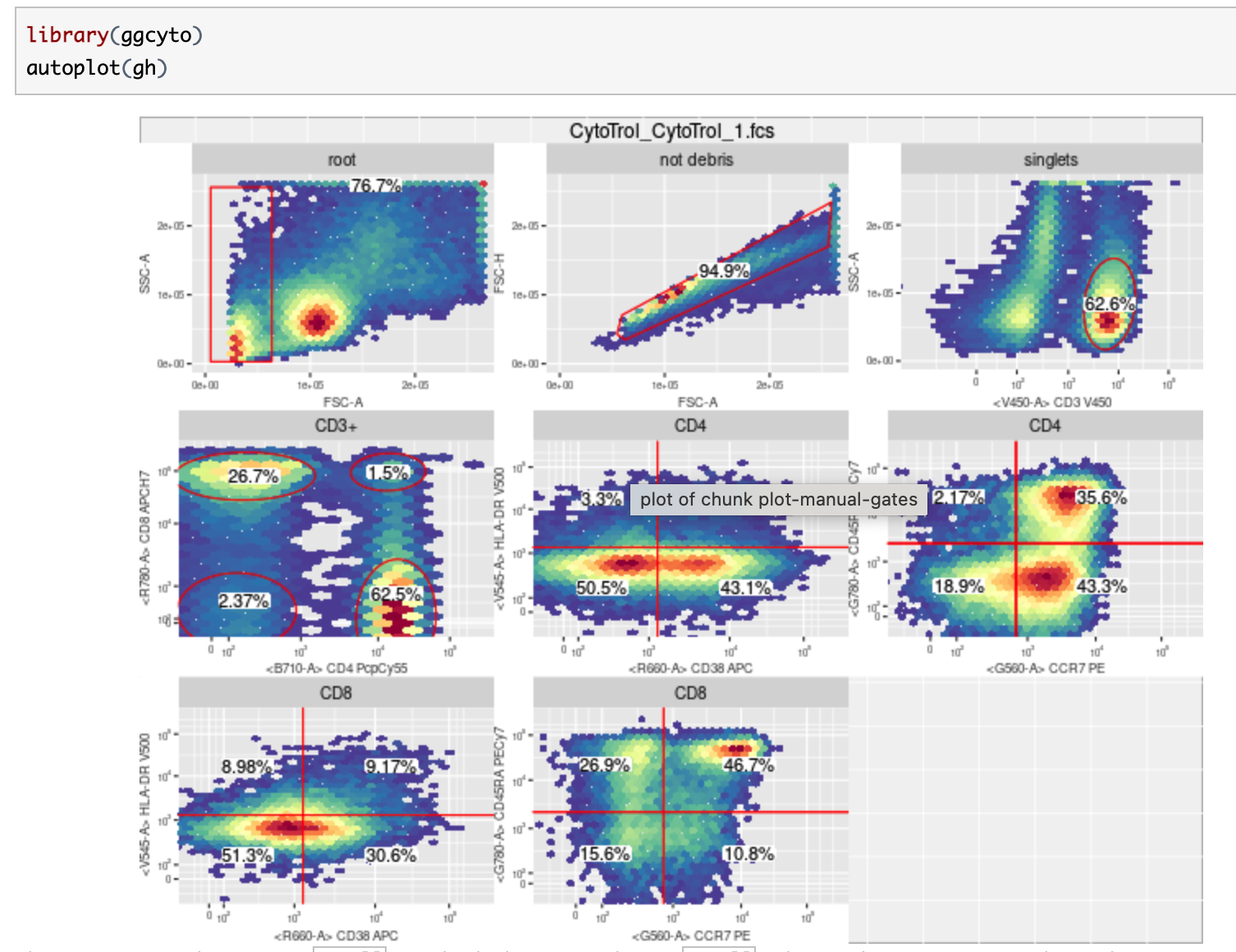
3、openCyto - An R package that providing an automated data analysis pipeline for flow cytometry
一个R包,为流式细胞术提供一个自动化的数据分析管道。
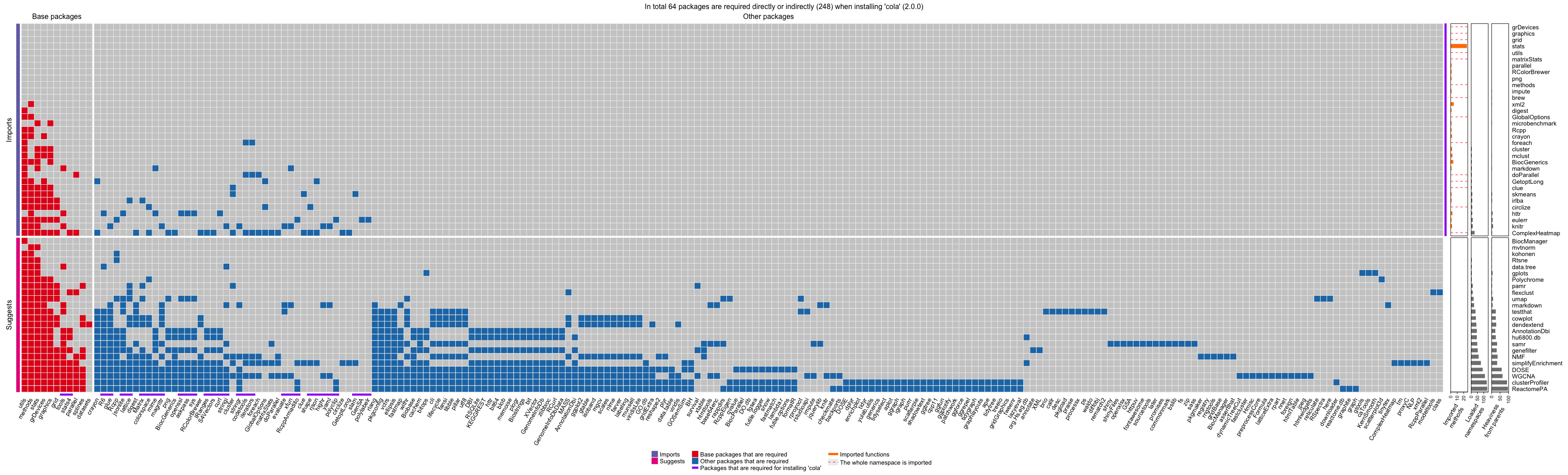
4、pkgndep - Analyzing Dependency Heaviness of R Packages
分析和可视化R包的依赖情况,对于开发和打包有用。
资源¶
1、Biology 723: Statistical Computing for Biologists
这是一个杜克大学的生物学课程。
2、Python Standard Libraries Cheatsheet
e.g.,
>>> import re
>>> strcmp = "www.baidu.com"
>>> re.match("www", strcmp).span() # span function to get index
(0, 3)
>>> re.match("baidu", strcmp) # re.match only match from the beginning of the string
>>> re.search("baidu", strcmp).span() # re.search search from all string and return the first
(4, 9)
>>> strcmp = "baidu.com/runoob.com"
>>> re.findall("com", strcmp) # re.findall find all results and return
['com', 'com']
>>> re.findall("b(.*?).", strcmp)
['', '']
>>> re.findall("b(.*?)c", strcmp)
['aidu.', '.']
赞赏¶
如果你想要支持本周刊,可以对推文进行赞赏或者提供的支付宝/微信二维码打赏。
感谢以下读者往期的赞赏:
- 李浩
- 祥子
订阅¶
这个周刊每周日发布,同步更新在微信公众号「生信协作组」(elegant-r)上。
微信搜索“生信协作组”或者扫描二维码,即可订阅。

(完)