生信爱好者周刊(第 148期):AI 革命不应该比喻为互联网,而是晶体管¶
这里记录每周值得分享的生信相关内容,周日发布。
本杂志开源(GitHub: openbiox/weekly),欢迎提交 issue,投稿或推荐生信相关内容。
封面图¶

本周话题:AI 革命不应该比喻为互联网,而是晶体管¶
文中回顾和精心编理了对OpenAI CEO Sam Altman的专访。Sam Altman认为,“AI具有令人难以置信的扩展性,迅速渗透到了各个领域。如今你使用的产品和服务背后都包含着大量的晶体管技术,但你不会把这些公司看作是’晶体管公司’。”
@Wangcy-rachel:目前,AI革命正迅速嵌入到我们的生活和各个行业中,AI会像晶体管一样将迅速迭代发展,并迅速应用到各个行业的产品中,这些产品将会极大方便我们的生活和工作。
生信研究¶
1、Nat Commun | 发表迄今最大的亚洲多祖先SV图谱,揭示亚洲人群的基因组变异模式
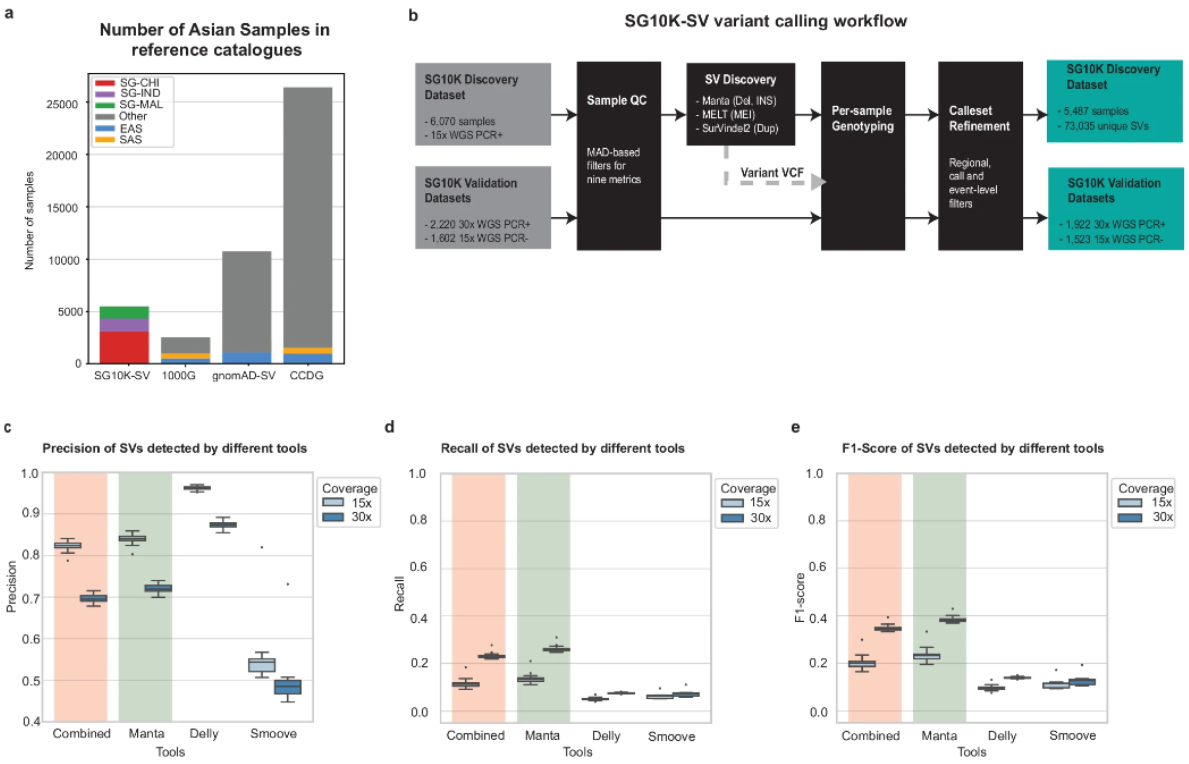
这篇文章是关于“Nat Commun发表迄今最大的亚洲多祖先SV图谱,揭示亚洲人群的基因组变异模式”的文献摘要,主要介绍了新加坡科技研究局联合多单位研究人员在Nature Communications上发表的一项大规模亚洲人群多祖先SV研究,该研究填补了亚洲人群基因组变异图谱的关键空白,对理解亚洲人群的基因组变异模式具有重要意义。
- 原文链接:https://www.nature.com/articles/s41467-024-53620-8
2、Nature Methods | 新算法利用空间转录组数据构建肿瘤的“空间进化图谱”
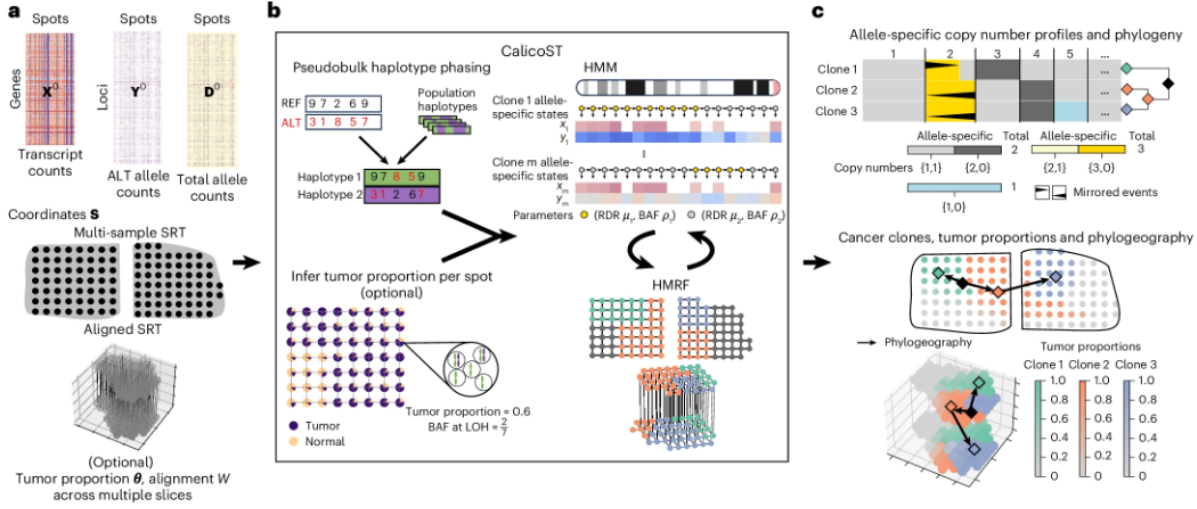
这篇文章是关于“Nature Methods发表新算法CalicoST,利用空间转录组数据构建肿瘤的‘空间进化图谱’”摘要,主要介绍了美国普林斯顿大学和华盛顿大学的科研团队合作开发的一种新算法CalicoST,该算法能从空间转录组数据中推断等位基因特异性拷贝数变异(CNAs),并重建肿瘤的空间进化轨迹。
- 原文链接:https://www.nature.com/articles/s41592-024-02438-9
3、Nature | 临床功能蛋白质组学分析策略解析胰腺癌细胞间信号转导网络
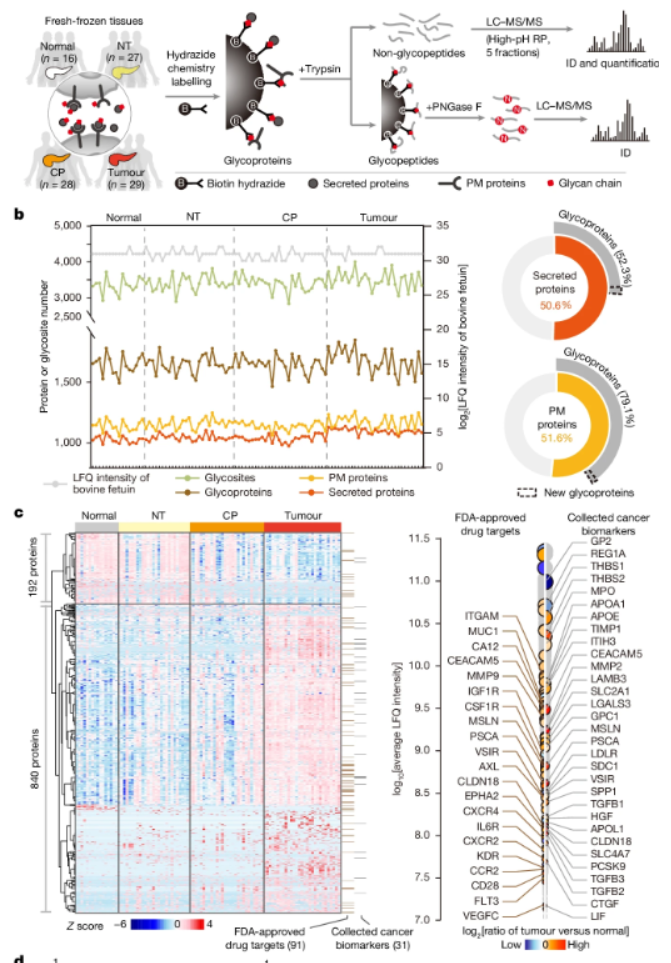
该篇Nature文章开发了多维度临床功能蛋白质组学分析策略并应用于系统解析胰腺癌肿瘤微环境中的细胞间信号转导网络,为发现新的胰腺癌标志物和药物靶点提供了新的思路和系统的功能蛋白质组大数据。
- 原文链接:https://www.nature.com/articles/s41586-024-08225-y
博文咨询¶
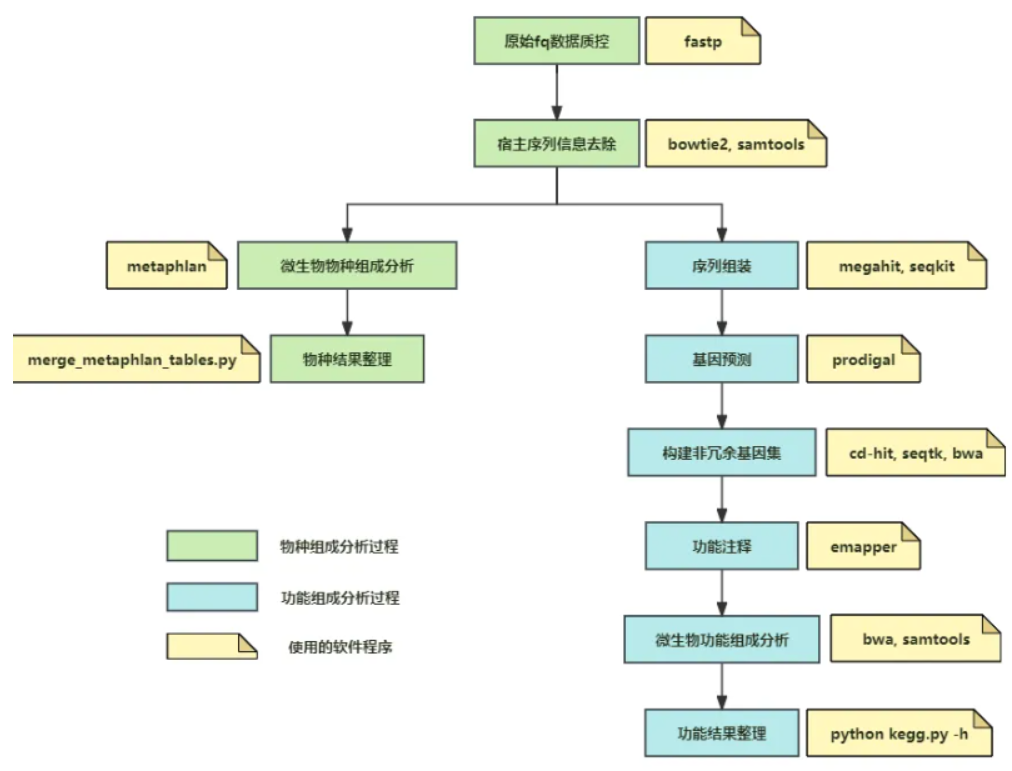
在人体肠道微生态研究领域,宏基因组测序技术是一种高效的研究手段,可以快速获取微生物群的物种特征以及其发挥的功能信息。目前有很多封装好的宏基因组分析软件或者包,但是对于初学者来说,直接利用这些封装好的一站式的软件或者包,对于理解宏基因组的分析过程不是很友好,有很多“黑箱”内容没办法理解。推荐的这篇文章描述了宏基因组分析过程的每个步骤的理解内容,适合初学者用来理解和学习。
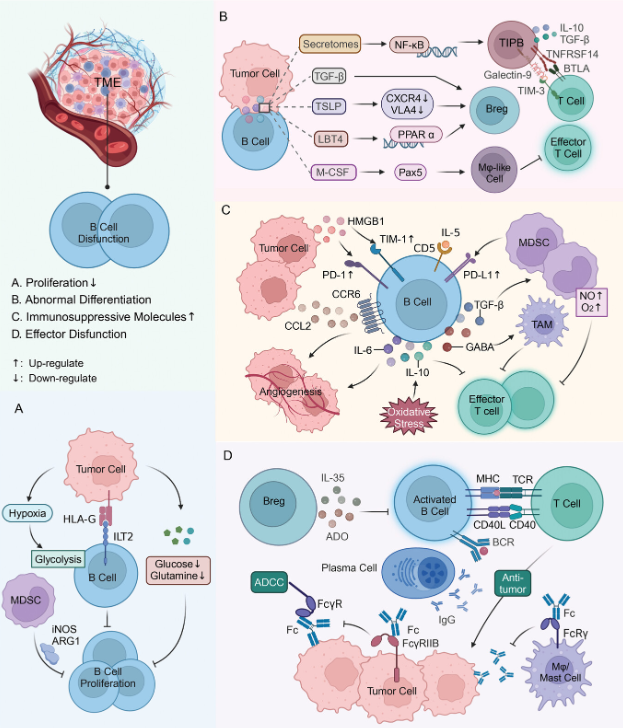
文中介绍了一篇有关B细胞免疫检查点与癌症治疗的综述,可以帮助我们了解B细胞免疫检查点与癌症治疗的详细知识和前沿热点。
- 原文链接:https://doi.org/10.1002/advs.202403423
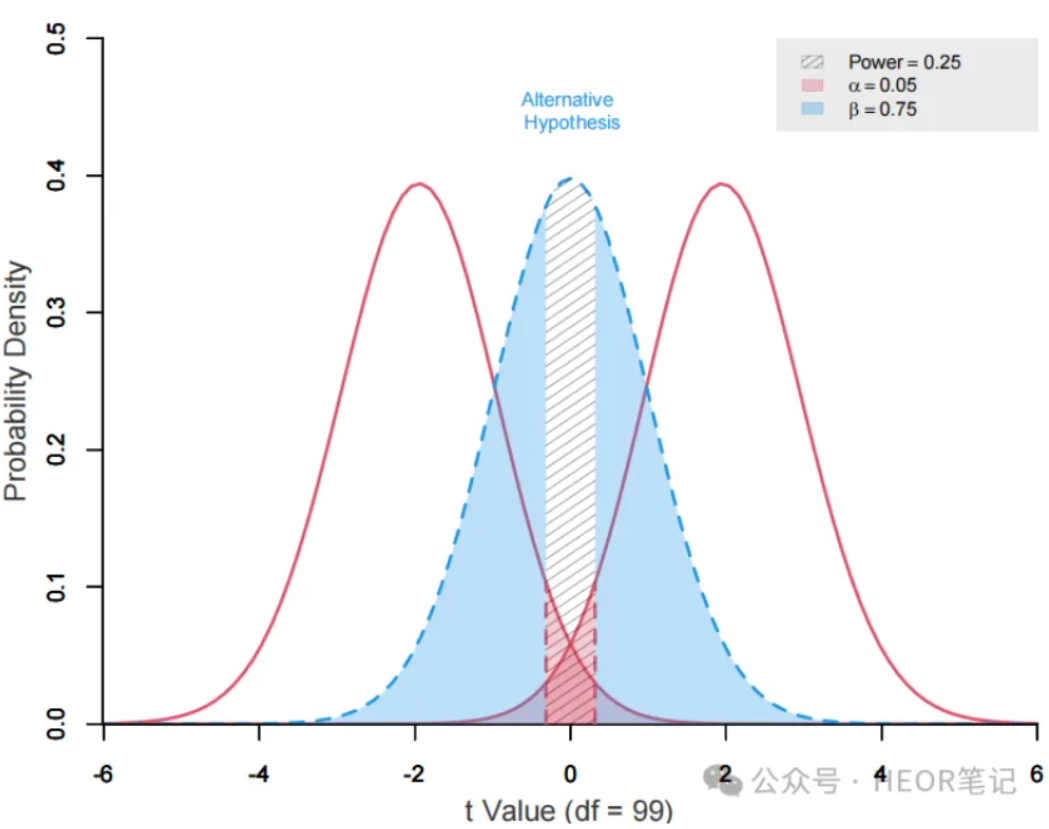
文中详细总结了使用R包pwrss计算各种检验和回归的样本量的代码以及示例图。
工具¶
7、Tidyplots | 让生命科学家轻松实现基于代码的数据可视化
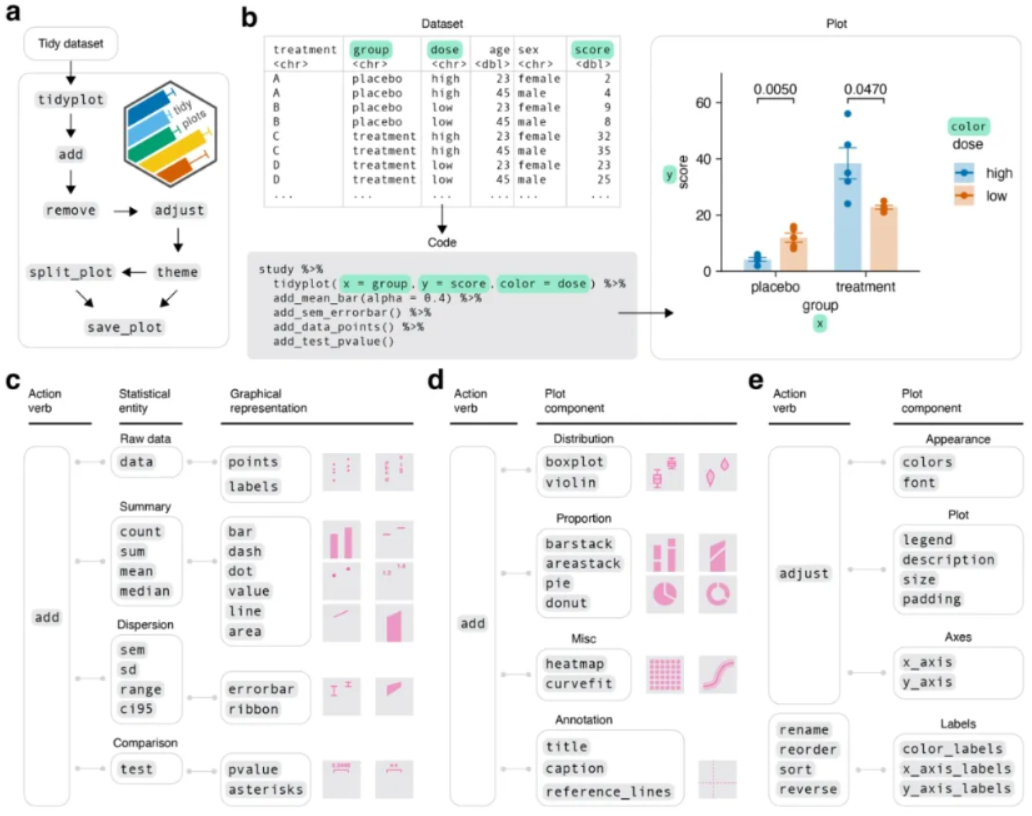
文中介绍了一个用户友好的基于代码的工具tidyplots,可用于创建可定制的、有洞察力的图表。
- 工具: https://github.com/jbengler/tidyplots
8、SplitsTree App | 使用系统发育树和网络进行交互式分析和可视化
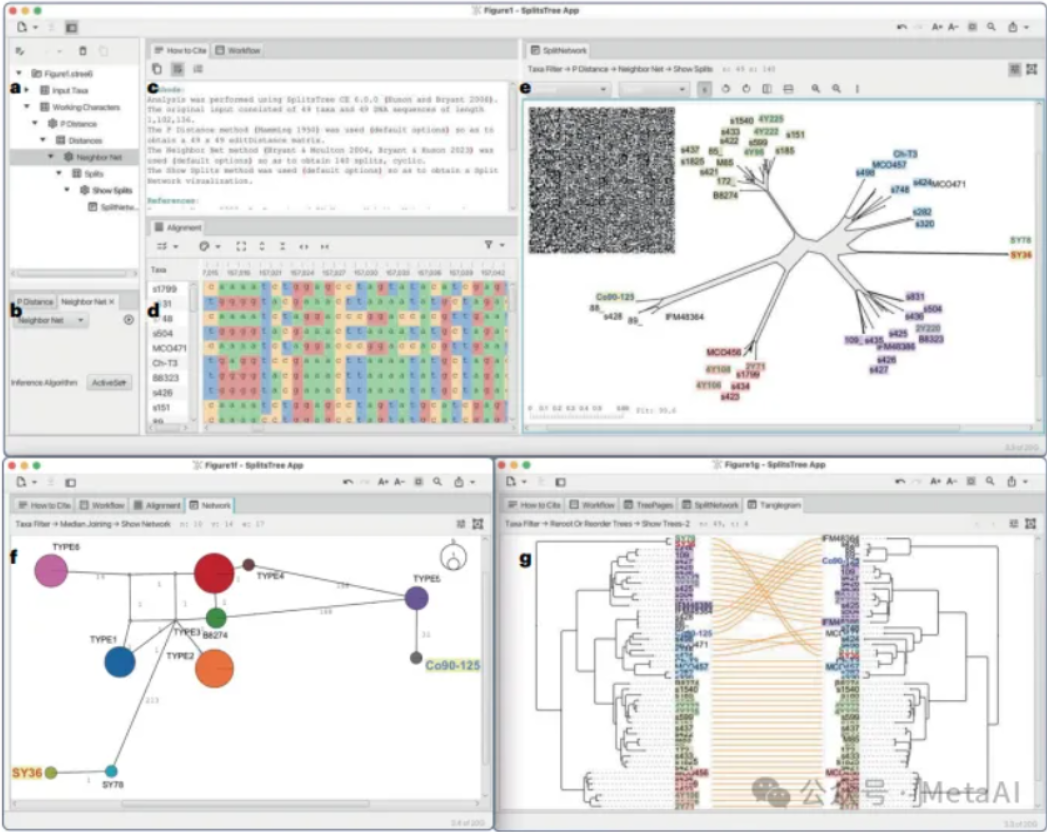
文中介绍了一个通过系统发育树和网络分析进化数据的软件SplitsTreeApp,集成了超过100种算法,并且能够处理各种输入格式,满足不同类型的数据分析需求。
- 工具:https://github.com/husonlab/splitstree6
9、DHR | 比blast和hmmer快几万倍同源基因鉴定新工具
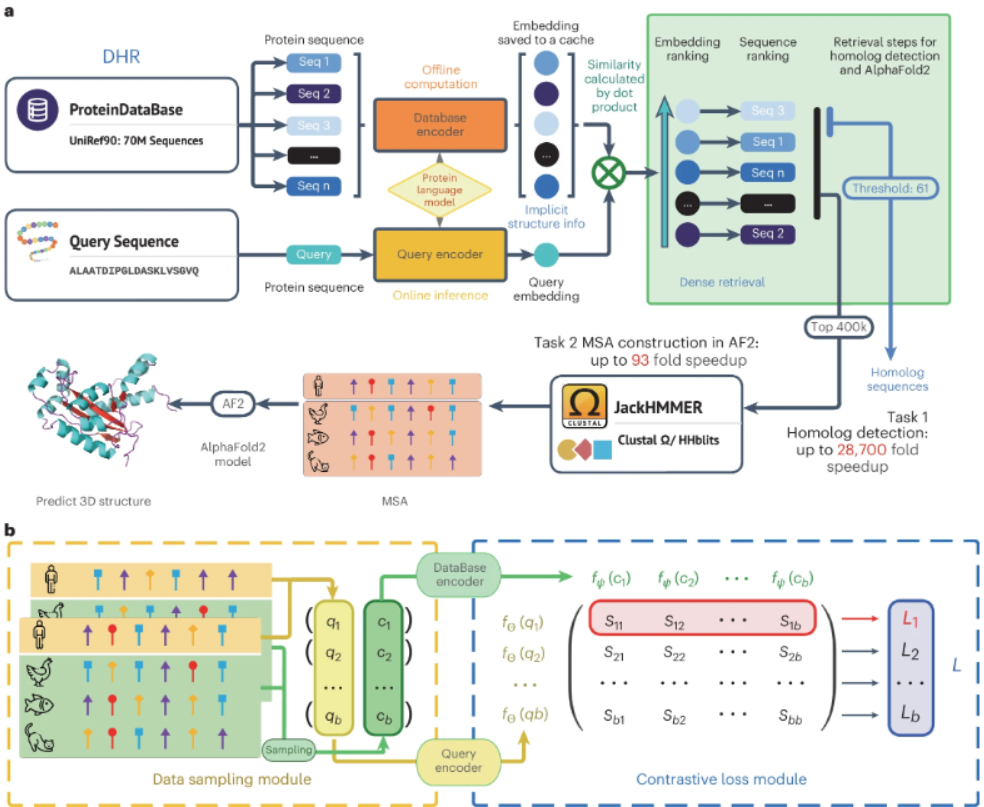
本文提供了一种超快速、高灵敏度的方法——密集同源物检索器(Dense Homolog Retriever, DHR),基于蛋白质语言模型和密集检索技术来检测同源物。它的双编码器架构为同一蛋白质序列生成不同的嵌入,并通过对比较这些表示来轻松定位同源物。其无需对齐的特性提高了速度,而蛋白质语言模型则在DHR嵌入中纳入了丰富的进化和结构信息。与以往的方法相比,DHR在灵敏度上提高了超过10%,在超家族级别对于难以使用基于对齐方法识别的样本,灵敏度提高了超过56%。它比传统方法如PSI-BLAST和DIAMOND快22倍以上,比HMMER快28,700倍以上。DHR独特发现的新的远缘同源物对于揭示已知蛋白质之间的联系以及提高我们对蛋白质进化、结构和功能的认识非常有用。
- 论文链接:https://www.nature.com/articles/s41587-024-02353-6
- 工具:https://github.com/ml4bio/Dense-Homolog-Retrieval
资源¶
10、AIGC算法必读论文清单

文中详细汇总了不同AIGC算法的必读论文、相关资料以及链接的清单,值得相关专业的朋友们阅读。
可以在 https://github.com/exacity/deeplearningbook-chinese/releases/download/v0.5-beta/dlbook_cn_v0.5-beta.pdf 下载 PDF。
贡献者(GitHub ID)¶
「Openbiox 生信周刊」运维小队:
@ShixiangWang(王诗翔)@kkjtmac(阚科佳)@NiEntropy(赵启祥)@He-Kai-fly(何凯)@JnanZhang(张佳楠)@Tomcxf(陈啸枫)@wangdepin(王德品)@kongjianyang(空间阳)@donghongyu2020(董弘禹)@DrRobinLuo(罗鹏)@Wangcy-rachel- 王春阳@zoe3251- 舒晨阳
订阅¶
这个周刊每周日发布,同步更新在微信公众号「生信协作组」(elegant-r)上。
微信搜索“生信协作组”或者扫描二维码,即可订阅。
(完)