生信爱好者周刊(第 127 期):高校“青椒”之死:困在“非升即走”里的海归博士¶
这里记录每周值得分享的生信相关内容,周日发布。
本杂志开源(GitHub: ShixiangWang/weekly),欢迎提交 issue,投稿或推荐生信相关内容。
封面图¶

本周话题:高校“青椒”之死:困在“非升即走”里的海归博士¶
无论是教学还是研究,没有考核当然也不可能。但需要明确的是,哪怕严格考核的大方向没错,也不意味着制度不可以完善。 比如有人指出,很多高校的考核标准过于严格、单一,又缺乏充分客观的同行评议机制,导致评价机制过于机械,将资质各异、禀赋不同的“青椒”们挤到了同质化的道路上。所以,如何进行人性化的改造,比如提高评价标准的多元性,扩展考核内容的承载量,提供一定的救济空间,保持严格与灵活的平衡,是当前应该探索的方向。
生信研究¶
1、 Nature communications | ASGARD:单细胞指导的药物再利用新策略
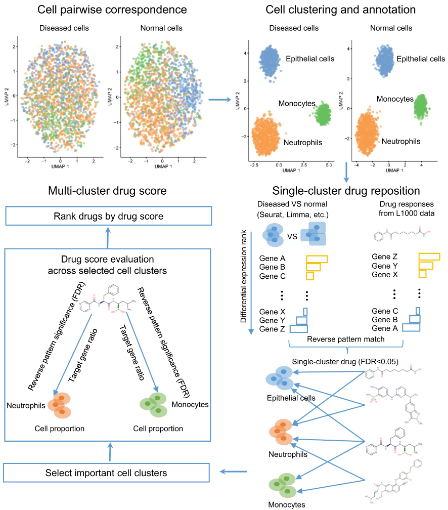
这期的nature communications上介绍了一种名为ASGARD(A Single-cell Guided Pipeline to Aid Repurposing of Drugs)的新工具,旨在利用单细胞RNA测序技术(scRNA-seq)来指导药物再利用,以实现精准医疗。ASGARD通过定义药物评分系统,考虑所有细胞亚群来推荐药物,从而应对每个患者内部的细胞异质性。研究发现,ASGARD在单药治疗的平均准确性上显著优于传统的基于群体细胞的两种药物再利用方法,并且在细胞群落水平的预测方法中表现更佳。通过使用TRANSACT药物反应预测方法对三阴性乳腺癌患者样本进行验证,发现许多排名靠前的药物已经获得FDA批准或正在进行对应疾病的临床试验。总体而言,ASGARD是一个有前景的药物再利用推荐工具,它为个性化医疗提供了由单细胞RNA测序指导的新策略。
- 论文链接:https://www.nature.com/articles/s41467-023-36637-3 工具链接:https://github.com/lanagarmire/ASGARD
2、Nat Med|基于22种常见癌症NGS数据开发AI模型预测治疗反应
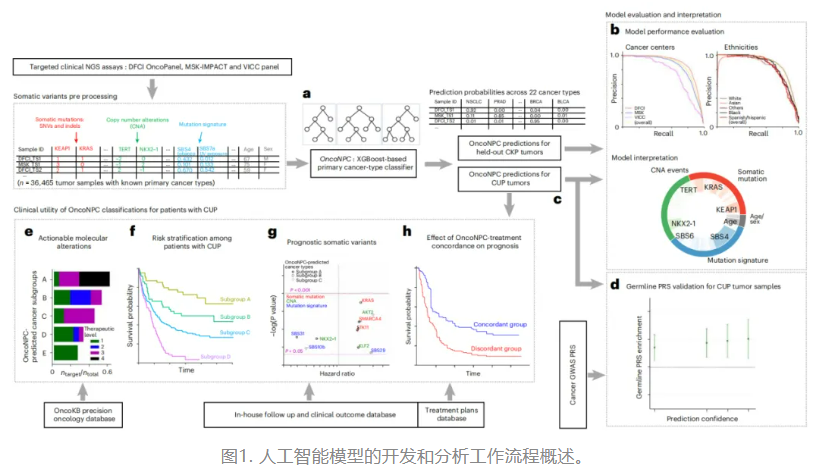
该研究开发了机器学习模型OncoNPC,可对肿瘤样本进行分类,并在已公布的多中心测试数据中提供了可靠且可解释的预测。研究团队观察到,通过OncoNPC预测方式进行治疗的CUP患者的生存期明显长于以不一致方式治疗的患者。OncoNPC极大地方便了个性化治疗的实施,对医生为CUP患者制定合适、精准的治疗策略具有重要意义。
- 论文链接:https://www.nature.com/articles/s41591-023-02482-6
3、Cancer Research丨绘制癌症中蛋白质的遗传调控位点图谱并揭示蛋白质丰度与药物响应和免疫浸润的关联

该研究利用癌症基因组图谱(TCGA)和癌症蛋白质组图谱(TCPA)中超过7000个癌症样本的数据进行了关联分析,对31种人类癌症的蛋白质遗传调控位点进行了鉴定,最终获得了超过10万个癌症pQTL。随后,作者进一步结合了癌症患者的临床数据分析了这些pQTL与患者总生存率的相关性,最终发现了超过8000个与患者总生存率关联的pQTL,这意味着这些变异位点可能通过影响特定蛋白质的表达,进而对癌症患者的预后产生影响 - 论文链接:https://doi.org/10.1158/0008-5472.CAN-23-0758
4、Nature Communications | 基因组变异解码框架Huatuo
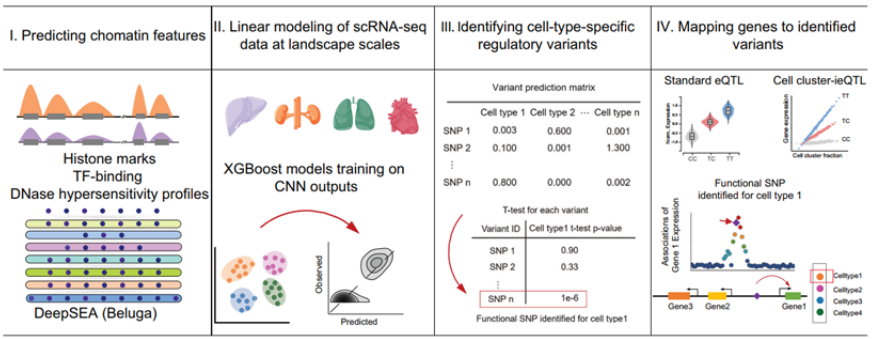
该研究提出了一种新颖的分析框架Huatuo(华佗),可仅依据来自少量生物学个体的基因表达谱,实现对突变转录效应在细胞类型水平上的推断,为解析复杂的基因表达遗传调控机制提供了一种高效途径。该研究进一步构建了一个全面的细胞类型特异性基因调控遗传变异景观,并通过各种基准测试验证了分析结果的合理性。最后,团队探索了基因组遗传变异与复杂表型之间的关系,为复杂性状和疾病的驱动细胞类型和因果变异提供了系统性见解。 - 论文链接:https://www.nature.com/articles/s41467-023-39538-7
博文资讯¶
deepin 开发团队宣布,在已经发布的 deepin V23 beta 版本中,现已正式适配 WSL。团队称他们选择了基于 WSL2 的适配方案,因为 WSL2 提供了 WSLg 扩展,可以提高性能,增强系统调用兼容性,也能让用户在 Windows 上享受 deepin 的美观界面和强大功能。目前看来,deepin WSL 还处于测试阶段,存在一些性能、兼容性、稳定性等方面的问题或不足。团队表示正在对应用进行更多的测试,以便发现并解决问题,提升用户体验。
6、75岁Hinton中国大会最新演讲「通往智能的两种道路」
Hinton 本次的演讲题目为「通往智能的两种道路」(Two Paths to Intelligence),即以数字形式执行的不朽计算和依赖于硬件的可朽计算,它们的代表分别是数字计算机和人类大脑。演讲最后,他重点谈到了大型语言模型(LLM)为他带来的对超级智能威胁的担忧,对于这个涉及人类文明未来的主题,他非常直白地展现了自己的悲观态度。
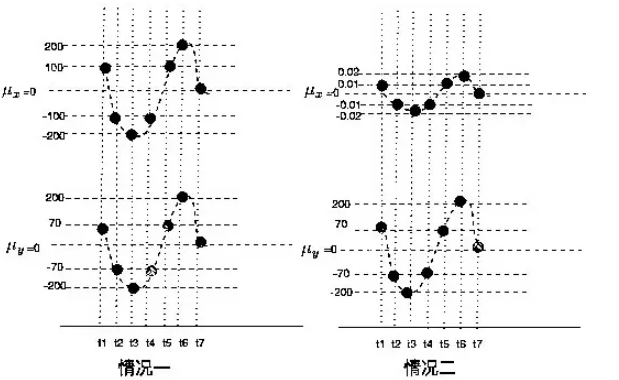 本文通过图文并茂的形式介绍了 2 个基本的统计概念。
本文通过图文并茂的形式介绍了 2 个基本的统计概念。
工具¶
 锐竞公司开发了许多有用的学术工具,这篇文章介绍了一个在线文献管理工具的使用。
锐竞公司开发了许多有用的学术工具,这篇文章介绍了一个在线文献管理工具的使用。
- 工具链接:https://scholar.rjmart.cn/#/?tg=RK4W&tab=2
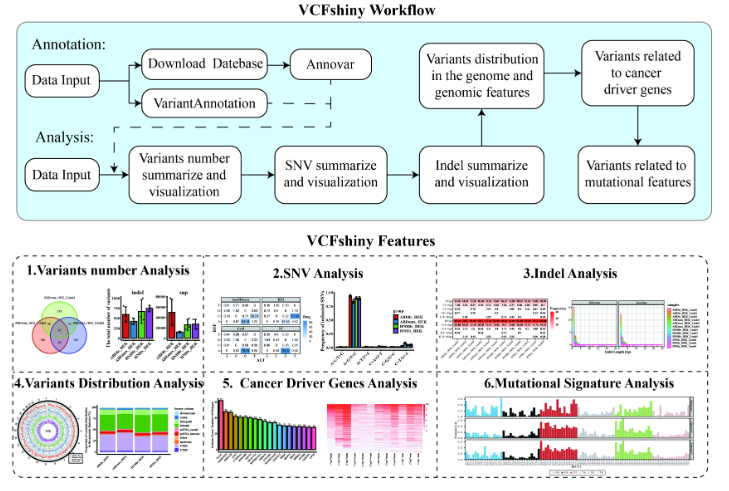
二代测序产生的变异通常以变异检出格式 (VCF) 文件重新编码。该VCF文件存储变异的详细信息,包括染色体位置,碱基序列,碱基质量,读取深度,基因型等。然而,对于没有生物信息学和编程背景的研究者来说,从VCF文件中破译相关的生物学见解可能是一项具有挑战性的任务。作者基于此开发了一个名为 VCFshiny 的 R/Shiny 应用程序包,用于以交互式和用户友好的方式解释和可视化存储在 VCF 文件中的各种变异。VCFshiny 能够汇总变异信息,包括总变异数、样本间变异重叠、单核苷酸变异的碱基改变、插入和缺失的长度分布、变异相关基因、基因组中的变异分布以及癌症驱动基因中的局部变异。在每次分析中,该软件提供了多种可视化方法,来获得用于发布和共享的图表。
- GitHub: https://github.com/openbiox/weekly/issues/2337
10、csvtk:表格处理神器
一个跨平台的生信数据处理小工具,可以节省大量写Python或者R脚本的时间
- Github: https://github.com/shenwei356/csvtk
ShellCheck是一个用于分析bash/sh Shell脚本的工具,旨在识别语法错误、语义问题以及潜在的陷阱。它提供了多种使用方式,包括网页、终端、编辑器集成以及构建或测试套件中。
资源¶
12、方糖技术
一个关于计算机内容且比较系统入门学习的博客
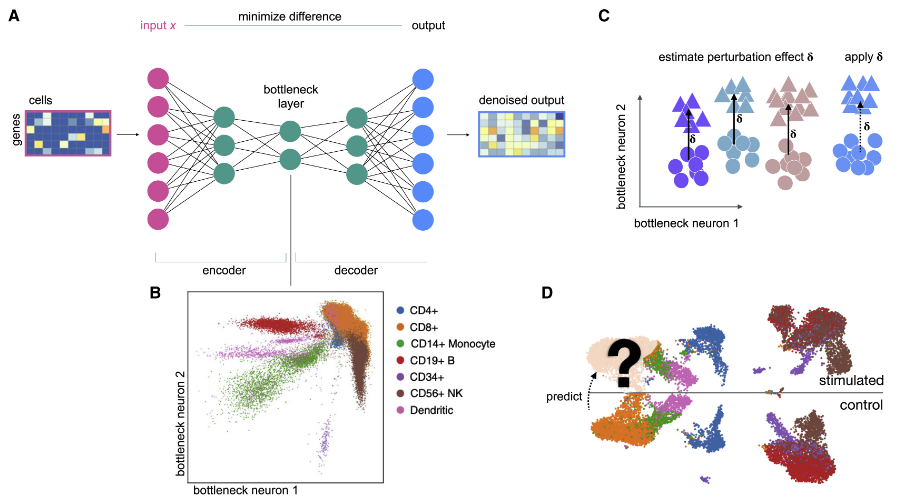
sc-pert是一个公共资源库,提供了一系列用于单细胞扰动组学(如CRISPR筛选和药物扰动)的模型和数据集。该资源库最初为2021年Cell system杂志的文章所策划,包含了多个与单细胞扰动模型评估相关的公共数据资源链接,如GDSC、DepMap PRISM、Therapeutics Data Commons等,以及不同来源的单细胞扰动数据集,支持研究人员进行深入分析和模型开发。
14、Appsilon-R Shiny Demo Gallery
Appsilon基于Shiny演示分享了一系列漂亮且易于使用的Shiny应用程序模板。每个模板都是开源的,完全可定制。
历史上的本周¶
- 第86期:如何做亮眼的研究生?
贡献者(GitHub ID)¶
「Openbiox 生信周刊」运维小队:
@ShixiangWang(王诗翔)@kkjtmac(阚科佳)@NiEntropy(赵启祥)@He-Kai-fly(何凯)@JnanZhang(张佳楠)@Tomcxf(陈啸枫)@wangdepin(王德品)@kongjianyang(空间阳)@donghongyu2020(董弘禹)@DrRobinLuo(罗鹏)
订阅¶
这个周刊每周日发布,同步更新在微信公众号「生信协作组」(elegant-r)上。
微信搜索“生信协作组”或者扫描二维码,即可订阅。

(完)